- 986
- ۱۴۰۲/۰۲/۰۸ - ۱۲:۱۶
- 884 بازدید
شرح فصل و نکات ویژه: در این فصل به مفهوم تنوع ژنتیکی و روشهای بررسی آن میپردازیم. پروژهها و بانکهای مربوط به SNPها در این فصل شرح داده میشوند. درختهای فیلوژنی و روشهای رسم آنها در این فصل شرح داده میشوند. پیشنهاد مطالعاتی: کتاب ”روشهای آنالیز فیلوژنتیکی” نوشته ”سید محسن نساج حسینی” از انتشارات حقشناس کتاب جامع در زمینه تجزیه و تحلیل فیلوژنتیکی میباشد. ۲۶۸-فصل سیزدهم ۱-۱۳ تنوع ژنتیکی در جمعیتها تنوع زیستی میتواند در سطوح[…]
شرح فصل و نکات ویژه:
- در این فصل به مفهوم تنوع ژنتیکی و روشهای بررسی آن میپردازیم.
- پروژهها و بانکهای مربوط به SNPها در این فصل شرح داده میشوند.
- درختهای فیلوژنی و روشهای رسم آنها در این فصل شرح داده میشوند.
پیشنهاد مطالعاتی:
- کتاب ”روشهای آنالیز فیلوژنتیکی” نوشته ”سید محسن نساج حسینی” از انتشارات حقشناس کتاب جامع در زمینه تجزیه و تحلیل فیلوژنتیکی میباشد.
۲۶۸-فصل سیزدهم
۱-۱۳ تنوع ژنتیکی در جمعیتها
تنوع زیستی میتواند در سطوح جمعیتی، گونهای و ژنتیکی سازمان دهی گردد، اگرچه تنوع ژنتیکی در پایین ترین حد این سلسله مراتب قرار میگیرد اما بدون آن یک جمعیت قادر به تکامل و سازگاری با تغییرات محیطی نمیباشد. تنوع ژنتیکی بر بالاترین سطوح تنوع زیستی تاثیرگذار است. تنوع زیستی در سطح ژنتیک منبع طبیعی گونههای وحشی را مییابد که این برای فهمیدن مقدار تغییرپذیری ژنتیکی مهم است، ضمنا علاوه بر این از نشانگرهای مورفولوژیکی، بیوشیمیایی و مولکولی، برخی از تغییرات جالب فیزیولوژیکی را میتوان یافت. تا مدتهای طولانی شرح گوناگونی فقط مبتنی بر خصلتهای موفولوژیکی بود اما تنوع مورفولوژیکی عموما محدود بوده و خصوصیات جاندار ممکن است در تمامی مراحل تکاملی موجود واضح نباشد. همچنین ظاهر جاندار میتواند تحت تاثیر شرایط محیطی قرار بگیرد. امروزه مارکرهای ژنتیکی متعددی به عنوان راه کاری مکمل برای روشهای سنتی دست یابی به تنوع ژنتیکی،پیشنهاد شدهاند.
ابزارهای مولکولی با به کارگیری توانایی خود در شناسایی تنوع در سطح DNA اطلاعات ارزشمندی پیرامون تنوع، پدید آوردهاند. برای ارزیابی تنوع گونه ای، طبقه بندی صحیح افراد ضروری است. شناسایی واحدهای تاکسونومیک و گونههای در حال انقراضی که ساختمان ژنتیکی آنها نسبت به دیگر اقوامشان دارای برتری میباشد، در توسعهی استراتژیهای مناسب حفاظتی از اهمیت ویژه برخوردار است. در مطالعات جمعیتی ابزارهای مولکولی برای تشخیص اینکه آیا دو فرد از لقاح والدین یکسان به وجود آمدهاند یا نه و تشخیص ارتباط خویشاوندی بین افراد به کار میروند. از یک دیدگاه عملیتر تشخیص اینکه چه کسی با چه کسی تولید مثل میکند برای مدیریت جمعیتهای کوچک بسیار مهم است. علاوه براین توانایی تشخیص شارش ژنی، ورود (ازگونههای دیگر) و آلودگیای که در نتیجه آمیزش دوجنس مخالف با هم ایجاد میشود (آلودگی ژنتیکی) برای دستیابی به خلوص، بسیار حیاتی بوده و به موضوع اصلی در بحث پیرامون امنیت ارگانیسمهای تغییر یافته ژنتیکی تبدیل گشته است.
از دیدگاهی تنوع زیستی معنی تنوع حیات در سه سطح زیر است:
۱- تنوع گونه ای که شمار و انواع گیاهان و حیوانات مختلف را در مقیاس محلی، منطقهای و جهانی توصیف میکند.
۲- تنوع ژنتیکی که به معنی تنوع ژنتیکی موجود در درون جمعیتها یا گونههای گیاهان و جانوران است.
۳- تنوع اکوسیستم که مقصود از آن تنوع زیستگاهها، جوامع زیست شناختی و اکوسیستمهایی است که زیست کره را میسازند.
تعریف تنوع ژنتیکی
تکامل مولکولی و فیلوژنتیک مولکولی در مفاهیم بیولوژیکی تکامل، به عنوان ظهور فرمهای بیولوژیکی از دیگر فرمها به صورت انتخاب طبیعی تعریف میگردد. مکانیسمی که باعث تکامل میگردد موتاسیونهای ژنتیکی است که به طور خود به خودی رخ میدهند. گوناگونی ترکیب ژنتیکی افراد متعلق به یک گونه، تنوع ژنتیکی نامیده میشود. ژنها واحدهای اساسی وراثت هستند که مشخصات فردی را در انتقال از والدین به زادگان کنترل میکنند مثل شکل برگ درختان کاج. هر فرد از نظر مشخصه با دیگر عضوهای گونه خود اندکی تفاوت دارد زیرا هر فرد (جز دوقلوهای همسان و کلونها) ریخت ژنتیکی منحصر بهفردی دارد. تنوع ژنتیکی صفات در موجودات بسیار است، مثل رنگ چشم در انسان که می تواند از قهوهای تیره یا حتی سیاه تا آبی یا حتی بیرنگ متفاوت باشد. فرمهای مختلف یک ژن را آلل مینامند. پس صفت رنگ چشم در انسان چندین آللی است. براساس یک قاعده کلی، تنوع زیستی در جمعیتهای بزرگ تر بیش از جمعیتهای کوچکتر است. به همین منوال، تنوع ژنتیکی گونههایی که چندین جمعیت دارند بیش از تنوع ژنتیکی گونههایی است که جمعیتهای محدودی دارند. گوناگونی ژنتیکی بین جمعیتها را معمولاً با مشخص کردن مقدار ”ناجور تخمی” در هر جمعیت مشخص میکنند چرا که هنوز منابع لازم برا ی مشخص نمودن ژنوتیپ تک تک افراد را در اختیار نداریم. ناجور تخمی را با بررسی صفتهایی خاص (معمولاً آنزیمها) به این صورت که آیا چند آلل برای آن صفت در جمعیت وجود دارد، میسنجند. اگر تمام اعضای یک جمعیت الگوی ژنتیکی یکسانی را برای یک صفت خاص داشته باشند میگویند که آن جمعیت از نظر صفت یاد شده جور تخم است و اگر برای یک ویژگی خاص بیش از یک الگوی ژنتیکی در جمعیتی مشهود باشد، آن جمعیت را از نظر صفت مذبور، ناجور تخم به شمار میآورند. با افزایش ناجور تخمی، تنوع ژنتیکی زیاد میشود.
تغییرات زیستی مانند متیلاسیون منجر به ایجاد جهش میشوند. جهش در یک ژن یک آلل بوجود میآورد. برخی از جهشها تاثیر فنوتیپی خاصی ندارند مانند جهشهایی که در بخشهای غیر رمز کننده ژنوم رخ میدهد. اما برخی از آنها
۲۶۹-تنوع ژنتیکی
که روی عملکرد پروتئین یا عناصر تنظیمی ژنها تاثیر دارند، کشنده میباشند. در برخی دیگر، وقوع برخی از جهشها نیز یک مزیت تکاملی را بوجود آورده است. آلل هر ژن یک تاریخچه تکاملی مستقل داشته و فراوانی آن در جمعیتهای مختلف متفاوت است. بررسی تنوع آللی در یک جمعیت مبحثی به نام تنوع ژنومی (Genome Diversity) را مطرح میکند که در سطح مولکولی در توالیها منعکس میشود.
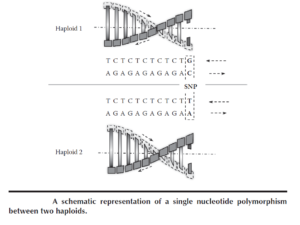
تصویر ۱-۱۳: نمایش شماتیک از یک SNP بین دو هاپلوئید.
جدول ۱-۱۳: دسته بندی انواع SNPها.

طرح HapMap

۲۷۰-فصل سیزدهم
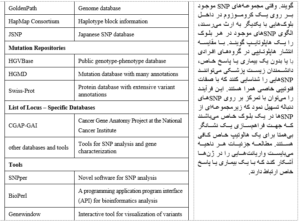
در سال ۲۰۰۲، با همکاری دانشمندانی از ایالات متحده، کانادا، چین، ژاپن، نیجریه و انگلستان طرح بینالمللی HapMap راهاندازی شد که یک تلاش جامع تعیین هویت SNPهای مرتبط با بیماریهای شایع انسانی و پاسخهای افتراقی به داروها میباشد. نقشه هالوتیپی (HapMap) میبایست منجر به تشخیص زودتر و صحیحتر شود و همچنین امید بهبود پیشگیری و مدیریت بیماران را به وجود میآورد.
بهعلاوه از شناخت مجموعه ژنتیکی فرد میتوان برای انتخاب داروها یا واکسنهای مطمئن و مؤثر در فرآیندی به نام فارماکوژنومیک استفاده کرد. این نشانگرهای ژنتیکی همچنین نشانههایی را فراهم میکنند که به واسطه آنها دانشمندان بتوانند در هنگام جستوجو و شناخت بیشتر فرآیندهای حیاتی وراثت و انتخاب ژنتیکی، ژنهای اختصاصی را جستوجو و تعیین هویت کنند.
۲-۱۳ تکنیکهای بررسی تنوع ژنتیکی

۲۷۱-تنوع ژنتیکی

ارزیابیهای مولکولی برای بررسی تنوع ژنتیکی بر دو روش کلی بررسی مارکرهای بیوشیمیایی و مارکرهای مولکولی تقسیمبندی میشود. در رابطه با بررسی توسط مارکرهای بیوشیمیایی به تحلیل پروتئینها و isozymeها میپردازند، الوآنزیمها آللهای متفاوت آنزیمها میباشند. با استفاده از این روش میتوان فرکانسهای آللی را به راحتی به دست آورد اما استفاده از این روش محدودهایتهای خاص خود را دارد از جمله این که واریانتهای آنزیمی بسیار محدود میباشند. بررسی توسط مارکرهای مولکولی خود به دو گروه کلی تقسیم میشود که به ترتیب تکنیکهای مبتنی بر PCR و تکنیکهای غیر وابسته به PCR (روشهای مبتنی بر هیبریداسیون) میباشند.
امروزه بیوانفورماتیک تحت عنوان ابزاری به منظور تجزیه و تحلیل دادهای زیستی در رابطه با مطالعات ژنومیکس، ترنسکریپتومیکس، پروتئومیکس و متابولومیکس در مسیر ایجاد یک سیستم بیولوژیک کارآمد کمکهای شایان ذکری را به دانشمندان بیوتکنولوژی داشته است که یکی از کارکردهای بیوانفورماتیک استفاده مستقیم آن در مطالعات فیلوژنتیکی و بررسی تنوع ژنتیکی میباشد. عموما برای دستیابی به اطلاعات ژنی و پروتئینی میتوان از بانکهای بیوانفورماتیکی استفاده کرد. امروزه شاهد هستیم بانکهایی تحت عنوان بانکهای ثانویه از بانکهای اولیه بیوانفورماتیک منتج شده اند که توسط آنها میتوان به صورت حرفهای به تجزیه تحلیل دمینهای پروتئینی به منظور بررسی تنوع آن در موجودات دیگر پرداخت. عموما در مطالعات تنوع ژنتیکی یک محقق به رسم نموداری درخت گونه تحت عنوان درخت فیلوژنی دست میزند تا از این طریق به شرح رابطه تکاملی موجودات بپردازد. از طریق این درختهای فیلوژنی میتوان به بررسی میزان تنوع ژنتیکی در جمعیت مورد نظر و مقایسه آن با سایر جمعیتها پرداخت، در ادامه شرح مختصری در رابطه با ابزارهای بیوانفورماتیکی برای رسم درخت فیلوژنی و الگوریتمهای آن خواهیم پرداخت. در جدول ۳-۱۳ روشها و مارکرهایی که اغلب مورد استفاده قرار میگیرند لیست شده اند. همچنین علامتهای اختصاری که معمولا برای نشانگرهای مختلف مولکولی استفاده میشود مشخص شدهاند.
روشهای آزمایشگاهی مختلفی برای نشان دادن SNPها وجود دارد که از آن جمله میتوان به Microarray،RFLP، TaqMan-RTPCR، Primer extesion، Oligo nucleotide Ligation اشاره کرد. یکی از مواردی که در تجزیه و تحلیل SNPها کاربرد دارد ارزیابی تاثیر هر یک از SNPها بر عملکرد پروتئین است. با توجه به تعداد زیاد دادههای SNP، از نظر تجربی بررسی تاثیر هر یک از SNPها بر عملکرد پروتئین غیرممکن است. بنابراین، محققان توسط روشهای محاسباتی به پیشبینی تغییرات اسید آمینه در عملکرد پروتئینها میپردازند. این روشها، همچنین به عنوان جایگزینی اسید آمینه
(amino acid substitution)یا AAS شناخته شده است.
۲۷۲-فصل سیزدهم
جدول ۴-۱۳: استراتژیهای مورد استفاده در AAS (Amino acid substitution).
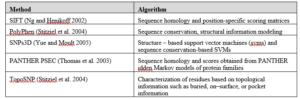
در جدول ۵-۱۳ ویژگیهای مختلف بعضی از تکنیکهای نشانگرهای مولکولی که غالبا در بررسی تنوع ژنتیکی استفاده میشود لیست شدهاند. سایر تکنیکها همچون Microarray نیز به صورت انبوه به بررسی پلیمورفیسمها میپردازد.
جدول ۵-۱۳: ویژگیهای بعضی از تکنیکهای نشانگرهای مولکولی.
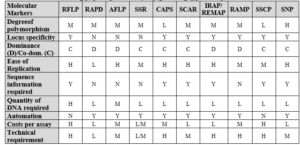
Key: H=High; M=Medium; L=Low; Y=Yes; N=No
۳-۱۳ پایگاه داده SNP
علیرغم نام SNP ، نوع جهش شامل جایگزینی تنها یک نوکلئوتید یا یک اسیدآمینه نمیشود، بلکه انواع دیگری از جهشها مانند اضافه شدنها و حذفهای کوتاه (Inseryions/Deletions or In/Del ) را نیز در برمیگیرد. اما به طور مشخص جهشهای ناشی از شکستهای بزرگ، بازآراییهای کروموزومی یا تغییر تعداد کروموزومها در این مقوله نمیگنجد و معمولا در بین دادههای سیتوژنتیک قابل جستوجو هستند. به طور طبیعی، بین دو جمعیت انسانی میانگین یک جهش به ازای ۱۲۵۰ باز وجود دارد. باید توجه داشت که توزیع تعداد جهشها در مناطق مختلف کروموزومی، در خلال جمعیتها و بین جمعیتها یا در موجودات مختلف یکسان نیست. همچنین بیشتر جهشها تاثیر فنوتیپی ویژهای ندارند و تنها آللهای یک ژن را بوجود میآورند. با استفاده از پروژه ژنوم انسان و توالی یابی آن SNPهای زیادی گزارش شده است که برای تشخیص بیماری مورد استفاده قرار میگیرند. کنسرسیوم SNP که توسط Lincoln Stein در Cold Spring Harbor مدیریت میشود تا سال ۲۰۰۸ تعداد ۱۸ میلیون SNP را در ژنوم انسان گزارش کرده است. دادههای جمعآوری شده توسط این کنسرسیوم از طریق پایگاه دادههای dbSNP در GenBank قابل جستوجو میباشد. در چنین پایگاههایی معمولا از IUPAC Code استفاده میشود که دانستن آنها در جایگزینی یک موقعیت با دو یا چند نوکلئوتید یا اسیدآمینه ضروری است.
۲۷۳-تنوع ژنتیکی
بانک اطلاعات جهشهای ژنتیکی انسان (HGMD) پایگاه داده مهم و مرکزی قابل دسترس جهت “جهشهای مرتبط با بیماری” میباشد. جهشهای خاموش که باعث تفسیر در اسید آمینه نمیشوند در این پایگاه ثبت نمیشوند مگر اینکه شواهد روش از مشارکت مستقیم آن در ایجاد یک بیماری خاص وجود داشته باشد. تغییرات پلیمورفیکی وارد شده در HGMD بهطورکلی یا در پروموتر ژن و یا در مناطق برنامهنویسی قرار دارند. با این حال باید توجه داشت که این SPNهایی که در خارج از این نواحی رخ میدهند ممکن است عواقبی برای بیان ژن، پردازش و … را دارا باشند.
جدول ۶-۱۳: IUPAC Code مربوط نوکلئوتیدها

جستوجو در پایگاه دادههای SNP
پایگاه دادههای dbSNP با وارد کردن کلید واژه (نام ژن) یا شماره دسترسی در پروژه ژنوم قابل جستوجو میباشد. این پایگاه داده از طریق وبسایت NCBI به آدرس www.ncbi.nlm.nih.gov در دسترس میباشد. با کلیک روی نام هر یک از SNPها میتوان به رکورد آن دست یافت.
۲۷۴-فصل سیزدهم
تصویر ۲-۱۳: نمایی از صفحه اول بانک SNP.
۴-۱۳ درخت فیلوژنی
فیلوژنتیک مطالعه تکاملی ارگانیسم زنده با استفاده از یک دیاگرام شبیه درخت است. شاخههای درخت واگرایی تکاملی را که فیلوژنی نامیده میشود را نشان میدهد. فیلوژنتیک میتواند از طریق یافتههای مربوط به فسیلها نیز مورد بررسی قرار بگیرد. خوشبختانه اطلاعات مولکولی که به شکل توالیهای DNA و پروتئین هستند نیز میتوانند چشم انداز خوبی از ارگانیسمها فراهم بیاورند چرا که تکامل ارگانسیم همراه با تکامل ماده ژنتیکی آنهاست. یکی از اهداف ساخت درخت فیلوژنتیک بر پایه توالی مولکولی،بازسازی تاریخچه تکاملی گونههای درگیر است. هرچند به طور محض،فیلوژنی ژنی (فیلوژنی استنباط شده از یک ژن یا توالی پروتئین) تنها تکامل ژن خاص یا پروتئین به رمز درآمده را توصیف میکند. این توالی ممکن است سریعتر یا کندتر از سایر ژنها در ژنوم نمو کند یا ممکن است تاریخچهی تکاملی متفاوتی از بقیه ژنوم داشته باشد. بنابراین،تکامل توالی ویژه ضرورتا با مسیر تکاملی گونهها هماهنگ نیست. تکامل گونهها نتیجهی ترکیبی از تکامل چندین ژن در ژنوم است. در یک درخت گونهها، نقطه شاخه شدن در گره داخلی نشانگر گونه زایی است، در حالی که در یک درخت ژنی گره داخلی مضاعف شدگی ژن را نشان میدهد. دو رخداد ممکن است همزمان باشند یا نباشند. بنابراین برای دسترسی به فیلوژنی گونهها، درختهای فیلوژنتیکی از خانواده ژنهای مختلف باید ساخته شوند تا تشخیص کلی در مورد تکامل گونهها داده شود.
در تجزیه و تحلیلهای فیلوژنتیک مولکولی به منظور نمایش تنوع ژنتیکی و مطالعات تکاملی نتیجه تحلیل به شکل یک درخت فیلوژنتیک نمایش داده میشود. این روابط تکاملی به وسیله مطالعه جهشهای جایگزینی، حذف، ازدیاد و … تعیین میشود که در معرض انتخاب طبیعی قرار میگیرند. در واقع فیلوژنی به مفهوم روابط تکاملی بین موجودات یا ژنهای آنها میباشد که در طول زمان با فرایندهایی از اجداد مشترک خود مشتق شدهاند. برای نشان دادن روابط فیلوژنتیک نیاز به رسم درختهای فیلوژنتیکی میباشد. امروزه درختان فیلوژنی براساس اطلاعات حاصل از توالیهای DNA و پروتئین ساخته میشوند تا اطلاعات مورفولوژیکی و یا حتی ایمونولوژیکی. سه دلیل عمده وجود دارد که باعث میشود تا درختان فیلوژنی رابراساس اطلاعات توالیهای نوکلئوتیدی و یا اطلاعات توالیهای آمینواسیدی رسم کنند:
۲۷۵-تنوع ژنتیکی
اول این که امتیاز دادن به توالیهای نوکلئوتیدی و آمینواسیدی سادهتر است و دوم اینکه هر نوکلئوتید یا آمینواسید را میتوان به عنوان یک خصوصیت برای بررسی روابط فیلوژنی در نظر گرفت، یعنی برای مثال با مقایسه دو توالی ۱۰۰۰ نوکلئوتیدی، ۱۰۰۰نقطه قابل مقایسه در دسترس است که در مقایسه با اطلاعات مورفولوژیکی بسیار زیاد است. در حقیقت دادههای مورفولوژیکی پتانسیل اطلاعاتی بسیار کمتری نسبت به دادههای مولکولی دارند و دلیل سوم اینکه توالیDNA و پروتئین، اطلاعاتی را هم برای حالتهای انشعاب یافته و هم برای حالتهای طبیعی در اختیار قرار میدهند.
۱-۴-۱۳ آشنایی با اصطلاحات درختهای فیلوژنی
خطوطی که در درخت هستند شاخه (Branch) نامیده میشوند. در راس شاخهها گونههای امروزه یا توالیهایی به نام taxa که به صورت منفرد taxon یا واحدهای طبقه بندی عملکردی قرار دارند. نقطه اتصال جایی که دو شاخهی مجاور به هم متصل میگردند گره (node ) نامیده میشود که نشان دهنده جد استنباط شده تاکسونهای موجود است. نقطه دو شاخه شدن در انتهای درخت گره ریشه است که جد مشترک همه اعضای درخت را نشان میدهد.
گروهی از تاکسونها که متعلق به شاخههای یکسانی هستند، به صورت یک clade یا گروه مونوفیلیتیک (Monophyletic) تعریف میشوند و یک خوشه (Cluster) نامیده میشوند. در یک گروه مونوفیلیتیک دو تاکسون یک جد مشترک را به اشتراک میگذارند. همچنین نسبت به هم تاکسونهای خواهری هستند (مانند تاکسای CوB در شکل). مسیر شاخه با رسم کردن ارتباط پایین آمدن جد روی درخت Lineage نامیده میشود، که معمولا با شاخهی درختی هم معنی است که منجر به گروه مونوفیلیتیک معین میشود. زمانی که تعدادی تاکسون در بیشتر از یک جد مشترک سهیم باشند، با تعریف clad مطابقت ندارند. در این حالت به صورت پارافیلیتیک (Paraphyletic) نامگذاری میشوند (مانند تاکسای B و C و D در شکل).
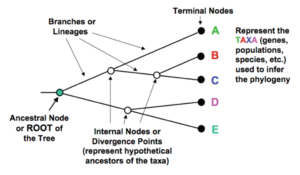
تصویر ۳-۱۳: بخشهای مختلف یک درخت فیلوژنی.
در یک درخت دو نوع گره میتواند وجود داشته باشد گرههای خارجی (Operating Taxonomic Unite = OTU) و گرههای داخلی (Hypothetic Taxonomic Unite= HTU).گرههای خارجی (انتهایی) یا تاکسون باقیمانده که “واحدهای تاکسونومیکی فعال” نامیده میشوند، یک واژه ژنتیکی است که میتواند انواعی از تاکسونهای قابل مقایسه (مانند خانواده موجودات، افراد یا استرینها از یک گونه خاص، یک سری ژنهای مشابه و یا حتی بخشهایی از ژنها) را نشان دهد مانند گرههای E, D, C, B, A در شکل بالا. گرههای داخلی نیز به نام “واحدهای تاکسونومیکی فرضی” نامیده میشوند و تأکیدی بر این است که آنها جدهای فرضی OTUها هستند مانند گرههایی که به صورت دایرههای توخالی در شکل بالا نشان داده شدهاند.
الگوی شاخهسازی در یک درخت، توپولوژی درخت نامیده میشود. زمانی که همه شاخهها روی درخت فیلوژنتیکی دو شاخه شدند به آن dichotomy میگویند. بعضی وقتها نقطهای روی درخت بیش از دو نسل دارد در نتیجه گره چند شاخهای تشکیل میشود.. فیلوژنی با شاخههای چند تایی شده را polytomy مینامند.
۲۷۶-فصل سیزدهم
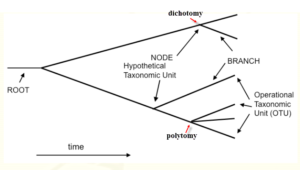
تصویر ۴-۱۳: درخت فیلوژنتیک دو شاخه و چند شاخه.
درخت فیلوژنتیکی میتواند ریشه دار و بدون ریشه باشد. برای درخت بدون ریشه جد مشترک نمیتوان فرض کرد. مسلما درخت ریشه دار نسبت به درخت بدون ریشه آگاهیدهندهتر است. برای برگرداندن درخت بدون ریشه به درخت ریشه دار ابتدا باید مشخص شود ریشه کجا است. دو راه برای تعیین ریشه وجود دارد. یکی از راهها استفاده از گروه بیرونی است که توالی آن با توالی مورد نظر همولوگ باشد اما از آن توالیها در زمان تکاملی زودتر جدا شده است مثلا استفاده از پرندگان به عنوان گروه بیرونی برای آنالیز پستانداران. اما در غیاب یک گروه بیرونی خوب درخت میتواند از طریق روش ریشه گرفتن نقطه میانی ریشه دار شود که در آن نقطه میانی دو گروه با بیشترین واگرایی به عنوان ریشه مشخص میشود. فرض میشود که در این نوع ریشه گرفتن،واگرایی از ریشه تا نوک درخت برای هر دو شاخه یکسان بوده و از نظریه ساعت مولکولی استفاده میشود.
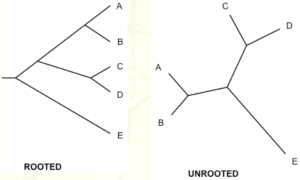
تصویر ۵-۱۳: درخت ریشه دار سمت چپ و درخت بدون ریشه سمت راست.
۲-۴-۱۳ ساعت مولکولی و برآورد زمان انشعاب توالیهای اجدادی
ساعت مولکولی (بر اساس فرضیه ساعت مولکولی) روشی است در تکامل مولکولی که از خصوصیات فسیلی و سرعت تغییرات مولکولی استفاده میکند تا زمان واگرایی (جدا شدن) دو گونه یا آرایه مختلف را در مقیاس زمان زمینشناسی استنتاج کند. این روش به منظور برآورد زمان وقوع گونهزایی و یا گسترش تکاملی استفاده میشود. دادههای مولکولی مورد استفاده برای اینچنین محاسباتی معمولاً توالی نوکلئوتیدی برای DNA و یا توالی اسید آمینه برای پروتئینها است. گاه به ساعت مولکولی نام ساعت ژنی یا ساعت تکاملی نیز میدهند. فرضیه ساعت مولکولی بیان میکند که برای هر ژن یا پروتئین، سرعت تغییرات مولکولی تقریباً ثابت است، یا به زبانی دیگر برای هر پروتئین یا ژن معین، نرخ تغییرات مولکولی تقریباً در هر رده تکاملی ثابت است.
۲۷۷-تنوع ژنتیکی
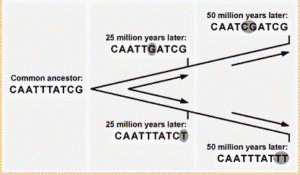
تصویر ۶-۱۳: این تصویر سرعت و نوع یک تغییر مولکولی فرضی را به نمایش گذاشته است.
هدف اولیهی ما از ترسیم درخت فیلوژنتیکی به دست آوردن روابط تکاملی بین توالیهای DNA است، که به وسیلهی توپولوژی درخت ترسیم شده به دست میآید. اما گاهی اوقات هدف ثانویهای مبنی بر کشف زمان انشعاب توالی اجدادی به توالیهای امروزی نیز دنبال میشود. البته هدف زمانی میسر میشود که ما از تاریخ تکاملی ژنهای آن موجود اطلاع داشته باشیم زمانی که ما اطلاعات حاصله از درخت ژن را در برابر اطلاعات حاصله در درخت گونهای قرار میدهیم، این جالب توجهتر خواهد بود، زیرا اکنون مشخص شده است که زمان انشعاب توالیهای اجدادی در حدود همان زمان حوادث گونه زایی است. برای اختصاص دادن زمان به نقاط شاخهای در درخت فیلوژنتیکی باید از ساعت مولکولی استفاده شود. فرضیه ساعت مولکولی ابتدا در دههی ۱۹۶۰ پیشنهاد شد و بیان کرد. که جایگزینی هر نوکلئوتید و یا جایگزینیهای آمینو اسید در توالیهای پروتئینی مورد مقایسه در یک میزان ثابت اتفاق میافتد. یعنی میزان اختلاف بین دو توالی را میتوان برای تخصیص زمان انشعاب آن دو توالی از توالی اجدادی بکار برد.
برای انجام این کار ابتدا باید ساعت مولکولی کالیبر شود تا بتوانیم میزان جایگزینی رخ داده به ازای هر میلیون سال را به دست آوریم برای مثال، فسیلها نشان میدهند که نزدیکترین جد مشترک انسان و اورانگوتان در ۱۳ میلیون سال پیش میزیسته است. برای کالیبر کردن ساعت مولکولی انسان توالیهای DNA انسان و اورانگوتان را با همدیگر مقایسه کرده تا مقدار جایگزینیهای نوکلئوتیدی که رخ داده اند را تعیین کرد. در عمل مشاهده شد که نمیتوان از ساعت مولکولی یک موجود برای همهی موجودات دیگر استفاده کرد. زیرا امروزه ثابت شده که ساعت مولکولی موجودات مختلف حتی ژنهای مختلف یک موجود با همدیگر متفاوت هستند. اختلافهای بین موجودات مختلف به خاطر زمان نسلی آنهاست. یعنی موجودات با طول نسل کمتر نسبت به موجودات با طول نسل بیشتر خطاهای همانند سازی بیشتری محتمل میشوند. بنابراین جوندگان، ساعت مولکولی تندتری نسبت به موجودات نخستین دارند. همچنین ساعت مولکولی در ژنهای میتوکندری سریعتر از ژنهای هستهای است. زیرا در میتوکندری ماشینهای تعمیر کمی نسبت به هسته وجود دارد. باید توجه داشت که سرعت ساعت مولکولی در طی ۱ تا ۲ میلیون سال گذشته افزایش یافته است. شاید به دلیل وجود جهشهایی است که به کندی در طی روند انتخاب طبیعی، حذف میشوند.
۳-۴-۱۳ اشکال نمایش درخت فیلوژنی
توپولوژی شاخهها در یک درخت ارتباط بین تاکسونها را مشخص میکند. درخت به روشهای مختلفی مانند کلادوگرام یا فیلوگرام میتواند ترسیم شود. در فیلوگرام طول شاخهها مقدار واگرایی تکاملی را نشان میدهد. این قبیل درختها مقیاسدار هستند که فایده آنها این است که که هم ارتباط تکاملی و هم اطلاعات در مورد زمان نسبی واگرایی شاخهها را نشان میدهند.
۲۷۸-فصل سیزدهم
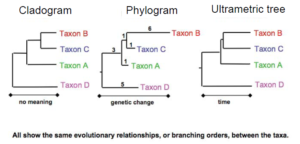
تصویر ۷-۱۳: درخت به روشهای مختلفی مانند کلادوگرام یا فیلوگرام میتواند ترسیم شود.
در کلادوگرام تاکسونهای خارجی در یک ستون یا یک ردیف به صورت مرتب به خط میشوند. طول شاخههای آن تناسبی با عدد تغییرات تکامل آن ندارد و به همین دلیل هیچ معنای فیلوژنتیکیای ندارد. در این نوع درختهای بیقیاس تنها توپولوژی درخت که ترتیب نسبی تاکسونها را نشان میدهد مهم است.
برای فراهمسازی اطلاعات توپولوژی درخت در برنامههای کامپیوتری فرمت نوشتاری ویژهای به عنوان فرمت Newick گسترش یافته است. در این فرمت درختها و تاکسونها در پرانتزهای تو در تو نشان داده میشوند. اگر درخت بیقیاس باشد طول شاخهها بعد از نام تاکسون میآید.
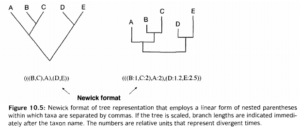
تصویر ۸-۱۳: فرمت Newick. در پرانتزهای داخلی تاکسا به وسیله کاما از یکدیگر جدا شدهاند.
۴-۴-۱۳ مراحل ساخت درخت به طور خلاصه
- انتخاب مارکر مولکولی
- همردیفی چندگانه بین توالیها
- انتخاب یک مدل تکاملی
- تعیین روش ساخت درخت
- ارزیابی قابل اطمینان بودن درخت
۲۷۹-تنوع ژنتیکی
۵-۴-۱۳ انتخاب مارکر
برای ساخت درخت فیلوژنتیک مولکولی میتوان هم از نوکلئوتید و هم اطلاعات توالی پروتئین استفاده کرد. انتخاب مارکرهای مولکولی مرحله مهمی است زیرا میتواند تفاوت زیادی در به دست آوردن یک درخت صحیح ایجاد کند. تصمیم این که از توالی نوکلئوتیدی یا پروتئینی استفاده شود بستگی به خصوصیات توالیها و اهداف مطالعه دارد. برای مطالعه ارگانیسمهای بسیار نزدیک به هم از توالیهای نوکلئوتیدی که سریعتر از پروتئین نمو میکنند میتوان استفاده کرد. برای مثال برای آنالیز تکاملی افراد مختلف در یک جمعیت اغلب از مناطق غیر کد کننده DNA میتوکندریایی استفاده میشود. برای مطالعه تکامل در گروههای واگراتر از توالیهای نوکلئوتیدی که به طور آهسته نمو میکنند مانند RNA ریبوزومی یا از توالی پروتئین میتوان استفاده کرد. اگر روابط فیلوژنتیکی در عمیقترین سطح باشند مثلاً بین باکتری و یوکاریوت استفاده از توالیهای پروتئینی حفاظت شده بهتر از توالیهای نوکلئوتیدی است.
توالیهای DNA گاهی اوقات بیشتر از توالیهای پروتئینی به خاطر استفاده ترجیحی کدون در ارگانیسمهای مختلف دچار سوگیری میشوند. در این مورد از کدونهای مختلف برای اسیدآمینههای یکسان استفاده میشود که برای تکامل قابل استناد نیست. همچنین با توجه به متفاوت بودن رمز کدونهای میتوکندریایی لازم است توالی DNA به پروتئین ترجمه شود. با توجه به فواید استفاده از توالی پروتئین توالیهای DNA میتوانند در مواردی مثل توالیهای مرتبط بسیار نزدیک آگهیدهنده باشند.
۶-۴-۱۳ همردیفی چندگانه توالیها
در فصل همردیفی توالیها به همردیفی چندگانه پرداخته شده است که یکی از الگوریتمهای رایج در این زمینه الگوریتم Clustal میباشد. در الگوریتم clustal برای هم تراز کردن چند توالی با هم سه گام اصلی وجود دارد. ۱)انجام هم تراز کردن جفتی. ۲) ساخت یک درخت فیلوژنتیک. ۳) استفاده از درخت فیلوژنتیک برای هم تراز کردن چندگانه توالیها. با انتخاب گزینهی “Do Complete Alignment” همهی این مراحل به طور خودکار انجام میشوند. گزینههای دیگر شامل
“Do Alignment from guide tree” و “Produce guide tree only” میباشد. کاربران میتوانند هم تراز کردن توالیها را با تنظیمات پیش فرض انجام دهند؛ هرچند معمولا با تغییر دادن تنظیمات با پارامترهای خاص خود نتیجهی بهتری بدست میآید. این پارامترها، جریمهی گشایش فاصله و جریمهی گسترش فاصله میباشند. نرم افزار دارای یک باکس میباشد که باید توالیهای نوکلوئیدی را با فرمت FASTA در آن قرار داد. این نرم افزار توالیها را با یکدیگر مقایسه میکند و بیشترین شباهت را اعلام میکند. در صفحهای که باز میشود کاربر میتواند با کلیک بر روی سربرگ Guide tree درخت رسم شده را مشاهده و تفسیر کند.
۷-۴-۱۳ انتخاب مدل جایگزینی
مدلهای آماری که برای تصحیح هموپلاژی استفاده میشوند، مدلهای جایگزینی یا مدلهای تکاملی نامیده میشوند. ساده ترین مدل جایگزینی نوکلئوتیدی مدل Jukes-cantor است که فرض میکند که تمام نوکلئوتیدها با احتمال مساوی جایگزین میشوند. یک مدل دیگر برای تصحیح فاصله تکاملی مدل دو پارامتری Kimura است. این مدل پیشرفته تر است و سرعت جهشهای انتقال و تبدیل در آن متفاوت فرض میشود که واقعبینانهتر است.
در زیستشناسی مولکولی و ژنتیک، جایگزینی انتقالی(Transition) به نوعی جهش نقطهای اطلاق میشود که طی آن یک نوکلئوتید پورین با یک نوکلئوتید پورین دیگر (A ↔ G) و یا یک نوکلئوتید پیریمیدین با یک نوکلئوتید پیریمیدین دیگر
(C ↔ T) جایگزین میشوند. از هر سه مورد چندریختی تک- نوکلئوتید، دو موردش «جایگزینی انتقالی» است.
۲۸۰-فصل سیزدهم
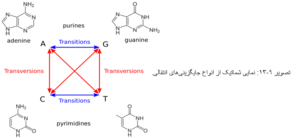
جایگزینی انتقالی را میتوان بوسیله «دِآمیناسیون اکسیداتیو» و توتومریزاسیون ایجاد کرد. با آنکه از لحاظ آماری، احتمال وقوع جایگزینی متقاطع باید بیشتر باشد، اما در عمل، «جایگزینی انتقالی» به میزان بیشتری در ژنوم یافت میشود که احتمالاً علت آن، مکانیسم ملکولی وقوع این پدیدهها است. بر اساس جایگزینیهایی که میتواند اتفاق بیفتد میتوانیم یک مدل برای فرگشت دی ان ای داشته باشیم بر این اساس تعدادی مدل مارکوف مختلف در مدل فرگشتی دیانای (Models of DNA evolution) مطرح شده است. این مدلهای جایگزینی به توضیح نوکلئوتیدهای جایگزین شده در فرگشت زیستی یک موجود زنده میپردازند. در تصویر زیر بر اساس سلسله مراتب الگوهای جایگزینی نوکلئوتیدی نمایش داده شدهاند. همانطور که در تصویر میبینید یکی از مدلهای جایگزینی جهت استخراج ماتریس فاصله مدل kimura میباشد.
مدلهای آماری دگرگونی نوکلئوتیدی جنبههایی از الگوهای تنوع را نشان میدهد که از فرایند تکامل حاصل میشود. الگوها بر اساس تعداد مولفههای مورد استفاده برای نمایش دگرگونی تکاملی، پیچیدگی مختلفی دارند. در حالی الگوهای ساده جایگزینیهای نوکلئوتیدی را به یک یا دو مولفه خلاصه میکنند، الگوهای دیگر میتوانند تا بیش از ۶۰ مولفه داشته باشند. مولفههای یک مدل میتواند اشاره به تفاوت در فراوانی نوکلئوتیدها، نرخ جایگزینی و تنوع میان موقعیتها داشته باشد. ترکیب مدلهای مختلف منجر به ایجاد الگوهای بسیاری شده است که چه بسا در یک یا چند مولفه مشترک باشند.
۲۸۱-تنوع ژنتیکی
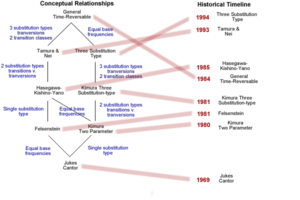
تصویر ۱۰-۱۳: مدلهای آماری دگرگونی نوکلئوتیدی.
۸-۴-۱۳ روشهای ایجاد و ارزیابی درختهای فیلوژنتیکی
درختهای فیلوژنتیک یا درخت تکامل نژادی (Phylogenetic tree) یک نمودار انشعابی ویا یک درخت است که روابط تکاملی در میان گونههای مختلف زیستی و یا حتی اشخاص را بر اساس شباهتها و تفاوتهای فیزیکی و یا خصوصیات ژنتیکی نشان میدهد taxaهایی که به هم در درخت متصل هستند، اشاره دارند به اینکه از یک جد مشترک جدا شدهاند. در یک درخت تکامل نژادی ریشهدار، هر گره نشان دهنده جد مشترکی برای فرزندان آن گره میباشد، و طول یالها در برخی از درختان ممکن است به عنوان تخمین زمان تفسیر شود. هر گره یک واحد طبقه بندی(taxonomic) نامیده میشود.گرههای داخلی عموما واحد طبقه بندی(taxonomic) فرضی (HTUs) نامیده میشوند و نمیتوان آنها را به طور مستقیم مشاهده کرد. این درختان در زمینههای زیستشناسی مانند بیوانفورماتیک، سیستماتیک وفیلوژنتیک مقایسهای کاربرد دارند.
روشهای ایجاد درختهای فیلوژنتیکی از دادههای مولکولی، نخست بر پایه این است که آیا روش مورد نظر حالت “ویژگی ناپیوسته”(discrete character) را به کار میبرد یا “ماتریس فاصلهای” (distance matrix) را و دوم بر پایهی این که آیا روش مورد نظر OTUها را به صورت تدریجی خوشه بندی میکند که “خوشه بندی تدریجی” (stepwise clustering) نامیده میشود و به تنها یک درخت که بهترین هم است، میانجامد یا تمام درختان که از نظر تئوری شدنی است را در نظر میگیرد که “جستوجوی فراگیر”(exhaustive search) نامیده میشود.
روشهای کاراکتری میتواند هر نوع از ویژگیهای ناپیوسته مانند ویژگیهای ریختشناسی، فیزیولوژیکی، نقشههای برشی یا دادههای توالی را به کار ببرند. بالعکس روشهای فاصلهای با محاسبهی اندازههای عدم تشابه هر جفت از OTUها در یک ماتریکس فاصلهی دو به دو آغاز میشود و سپس روابط فیلوژنتیکی OTUها را از ماتریکس محاسبه میکند. به نظر میرسد این روشها به خصوص برای بررسی توالیها بسیار مناسبند. مزیت اصلی روشهای فاصلهای اینست که آنها کمتر به رایانههای قوی نیاز دارند و هنگامی که تاکسونهای بسیاری مقایسه میشوند، مهم است.
۲۸۲-فصل سیزدهم
جدول ۷-۱۳: روشهای ایجاد درختهای فیلوژنتیکی بر اساس دو مولفه نوع داده و نوع آزمون ارزیابی درخت.
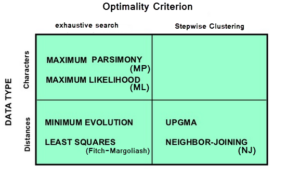
بر اساس این جدول روشهای “ماتریس فاصلهای” (distance matrix) و روشهای “ویژگی ناپیوسته” (discrete character) را Type of Data در نظر گرفته است و برای دستهبندی روشها بر اساس معیار بهینگی (Optimality Criterion) به دو دسته stepwise clustering و exhaustive search تقسیم شدند. بر اساس تقسیمبندی شرح داده شده روشهای MP, ML, NJ و … در بخش مناسب قرار گرفتهاند.
آزمونهای آماری متفاوتی برای ارزیابی درخت ترسیم شده وجود دارد، اما این آزمونها پیچیده هستند، زیرا درخت ترسیم شده بیشتر ژئومتریک است تا عددی. بنابراین دقت و صحت یک بخش از درخت ممکن است کمتر یا بیشتر از صحت بخشهای دیگر باشد. یکی از معروفترین روشها در این خصوص روش بوت استرپ است. که برای بررسی اطمینان نقاط شاخههای مختلف داخل یک درخت به کار می رود. این روش در سال ۱۹۷۹ به عنوان روش ارزیابی درختها در تجزیههای فیلوژنی به وسیلهی فلسنستین معرفی شد. در حقیقت، بوت استرپ یک آزمون آماری درختهای فیلوژنتیکی با استفاده از روش نمونهبرداریهای متعدد از دادههای اولیه است و برای آزمون درختهای ترسیم شده با روشهای فاصلهای و درستنمایی به کار گرفته میشود. نتایج حاصل از این آزمون به صورت عددی در کنار گره یک درخت نشان داده میشود که در واقع درصد دفعاتی است که هر شاخه مشخص در نمونهبرداری مختلف توسط این آزمون و درختهای حاصل از آن تشکیل میشود. روش bootstrap یک راه مفید برای برآورد اساس توزیع با نمونهگیری مجدد ازمجموعه دادههای اولیه است و در شرایطی به کار میرود که اساس توزیع نمونهگیری یا ناشناخته است یا برای نتیجهگیری تحلیلی مشکل است.
روشهای جستوجوی فراگیر (exhaustive search)، به منظور ارزیابی درخت میباشند که توپولوژیهای ممکن (به صورت نظری) درخت را برای تعداد معینی از تاکسونها آزمون میکنند که معیارهای معینی برای انتخاب بهترین درخت به کار میبرد. روشهای خوشه بندی تدریجی (stepwise clustering) معمولا سریع بوده و میتوانند تعداد زیادی از OTUها را با هم تطبیق دهند. بیشتر روشهای ماتریکس فاصلهای، خوشه بندی تدریجی را برای محاسبهی بهترین درخت به کار میبرند در حالی که بیشتر روشهای کاراکتری روشهای جستوجوی فراگیر را پذیرفتهاند.
روش حداکثر صرفهجویی (Maximum Parsimony=MP)
پیدا کردن توپولوژی درخت برای یک مجموعه توالیهای همردیف شده است که با کمترین تعداد تغییر کاراکتر (جهش) میتواند توضیح داده شود.
روش حداکثر درستنمایی (Maximum Linelihood=ML)
این الگوریتم احتمال مورد انتظار برای هر نوکلئوتید (یا اسید آمینه) ممکن در گرههای اجدادی (داخلی) را محاسبه میکند و درستنمایی ساختار درخت را از این احتمالات به دست میآورد. این روش با روش MP در آزمودن همهی توپولوژیهای قابل قبول مشابه است.
۲۸۳-تنوع ژنتیکی
روش UPGMA (Unweighted Pair Group Method with Arithmetic Mean)
قدیمیترین و سادهترین روش به کار رفته برای بازسازی درختان فیلوژنتیکی از دادههای فاصلهای میباشد. خوشه بندی با جستوجوی کوچکترین مقدار در ماتریس فاصلهای دو به دو انجام میشود.
روش اتصال مجاور (Neighbor Joining=NJ)
درخت را با یافتن پیدرپی جفتهای نزدیک به هم رسم میکند که جفت OTUها با یک گره داخلی به هم وصل میشوند.
روش FM (Finch Margoliash)
یک روش ماتریس فاصلهای است که همه درختان ممکن برای کوچکترین طول کل شاخه را ارزیابی میکند و یک الگوریتم اختصاصی به کار میبرد که فاصلههای دو به دو را در نظر میگیرد.
۹-۴-۱۳ نرم افزارهای رسم درخت فیلوژنی
در اینجا دو نرم افزار برای رسم درختهای فیلوژنی معرفی میشود که مورد اول نرم افزاری آنلاین میباشد که کاربر توالیها را به نرم افزار میدهد و نرم افزار درخت را رسم میکند و تنوع ژنتیکی را به نمایش میگذارد. نرم افزار دوم رایگان میباشد و قابل نصب بر روی کامپیوترهای خانگی است که در این نرم افزار با قابلیتهای بیشتر کاربر میتواند الگوریتم خاصی را برای رسم درخت فیلوژنی انتخاب کند.
Clustal
یکی از برنامههای آنلاین معروف برای هم تراز کردن چندگانه توالیها است. این نرم افزار توسط انستیتوی بیوانفورماتیک اروپا طراحی و مدیریت میشود و از طریق لینک http://www.ebi.ac.uk/Tools/msa/ در دسترس میباشد.
MEGA
یکی از نرم افزارهای پرکاربرد در زمینه رسم درختهای فیلوژنی نرم افزار MEGA میباشد. این نرم افزار به صورت رایگان قابل دانلود میباشد و با الگوریتمهای مختلف به رسم درختهای فیلوژنتیکی میپردازد. الگوریتمهای مختلفی در نرم افزارها به کار میرود و یا گاها مشاهده میکنیم در یک نرم افزار چند الگوریتم مختلف گنجانده شده است. الگوریتمهای مختلف بر اساس در نظر گرفتن فاکتورهای خاصی به حدس روابط فیلوژنتیک میپردازند. برای مثال میتوان از روشهای Parsimony Method Maximum و Neighbour-Joining Method برای مطالعه روابط سیستماتیک یا فیلوژنی در نرم افزار MEGA نام برد.
۲۸۴-فصل سیزدهم
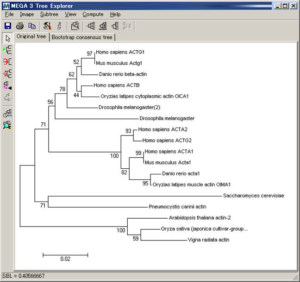
تصویر ۱۱-۱۳: نمایی از یک درخت فیلوژنتیک رسم شده توسط نرم افزار mega.
روش استفاده از نرم افزار MEGA طبق الگوی زیر میباشد
پس از دانلود نرم افزار (http://megasoftware.net) و نصب آن کاربر باید این مراحل را بپیماید. ۱: نرم افزار را اجرا کنید. ۲: وارد سربرگ aligment شوید. ۳: گزینه Aligment Texplorer /Clustal را انتخاب کنید. ۴: بر روی Ok کلیک کند (Yes برای DNA و No برای پروتئین میباشد). ۵: این مرحله یک مرحله مهم میباشد که به سه بخش تقسیم میشود. کاربر باید توجه کند هر کدام از بخشهای الف یا ب و یا ج را انتخاب کرد مد نظر داشته باشد که باید ادامه کار را از مرحله ۶ ادامه دهد.
الف: کاربر میتواند سربرگweb را باز کند با انتخاب گزینه Query Genbank توسط اینترنت توالی خود را پیدا کند و هر وقت به صفحه توالی مورد نظر رسید در نرم افزار بر روی add to aligment کلیک کند.
ب: در این مرحله به جای استفاده از اینترنت میتوان از سربرگ data گزینهretrieve sequnces from file را انتخاب و فایلهایی را که قبلا کاربر در پروژههای قبلی ایجاد کرده و یا پروژههای نمونهای را که در نرم افزار وجود دارد را اجرا کرد و یا به منظور آموزش از این پروژهها استفاده کند.
ج: اگر توالی مورد نظر را در دسترس داریم باید یک فایل با فرمت مگا بسازیم، به این صورت که بر روی دکمه Insert blank sequence در سربرگ edit کلیک کنیم تا یک سربرگ بسازیم، این کار را میتوانیم بر مبنای هر چند تا
۲۸۵-تنوع ژنتیکی
توالی که میخواهیم با یکدیگر مقایسه کنیم انجام دهیم و حال با کلیک بر روی هر کدام از این مربعها که sequnce1 تا n نام گرفتهاند میتوانیم توالی خود را قرار دهیم. بعد از اتمام کار میتوانیم یک فایل با فرمتMAS بسازیم. با کلیک بر روی create new data فایل MAS ساخته میشود.
۶: در این مرحله کاربر باید از سربرگ edit گزینه select all را انتخاب کند. ۷: بر روی سربرگ aligment کلیک کند و گزینه Align by ClustalW را انتخاب کنید. ۸: سپس با کلیک بر روی ok نتیجه مشخص میشود. ۹: از سربرگ data گزینه save باید انتخاب شود. ۱۰: در این مرحله کاربر باید از این صفحه خارج شود و از سربرگ data گزینه Exit Alnexplorer را انتخاب کند. کادر محاورهای باز میشود تحت عنوان save mega file که در اینجا بر روی yes باید کلیک شود و در کادر بعد بر روی save بایدکلیک شود و در مرحله بعد یک title میخواهد که یک نام با حروف انگلیسی باید به پروژه داده شود. در نهایت یک فایل با فرمت MEG ساخته میشود. ۱۱: حال دو صفحه جلوی کاربر باز است. باید کاربر به صفحه اصلی برود و در این زمان چند سربرگ جدید به صفحه اصلی اضافه میشود. از سربرگ phylogeny گزینه construct phlogeny و یا Bootstrap test میتواند انتخاب شود و در مرحله بعد میتوان روش Neighbor – joining (NJ) و سپس compute را انتخاب شود. حالا درخت فیلوژنی مورد نظر به صورت گرافیکی طراحی میشود و به کاربر ارائه میشود.
۵-۱۳ انتخاب طبیعی
گزینش طبیعی یا انتخاب طبیعی فرایندی است که در طی نسلهای پیاپی، سبب شیوع آن دسته از صفات ارثی میشود که احتمال زنده ماندن و موفقیتزاد ولد یک ارگانیسم را در یک جمعیت افزایش میدهند. اعضای یک جمعیت از موجودات زنده بهطور طبیعی متفاوتند. بسیاری از این تفاوتها اثری مستقیم روی توان ارگانیسم برای ادامه حیات ندارند، حال آنکه برخی از تفاوتها میتوانند تأثیری مثبت روی توان موجود زنده برای بقا داشته باشند. اگر چه انتخاب طبیعی بر روی صفات ظاهری (فنوتیپ) یک ارگانیسم عمل میکند اما در حقیقت ژن که بخش قابل توارث و پایه تمامی اینگونه صفات ظاهری ارگانیسم است، با افزایش شیوع خود در جمعیت، سبب افزایش شیوع اینگونه صفات مثبت انتخاب شده میگردد. با گذشت زمان، این فرآیند میتواند منجر به انطباق ارگانیسم با یک زیستگاه ویژه شده و ممکن است سرانجام به ظهور گونههای جدید بیانجامد. اگر چه انتخاب طبیعی تنها فرآیندی نیست که منجر به تکامل در درون یک جمعیت از موجودات زنده میشود اما باید آنرا یکی از مهمترین فرآیندهای اثرگذار دانست. با توجه به این تعاریف، برای مثال وجود دو آنزیم لیزوزیم مختلف با عملکردهای مختلف در گاو میتواند نشاندهنده انتخاب تکاملی مثبت باشد اما وجود برخی از لکهها در روی پوست یا تفاوت در رنگ پوست گاوها در یک منطقه جغرافیایی را نمیتوان انتخاب تکاملی مثبت یاد کرد. در حالی که، شاید تغییر رنگ پوست یک حیوان در قطب به رنگ سفید، یک انتخاب تکاملی مثبت باشد. همانطور که در ابتدای فصل ذکر شد؛ با افزایش ناجور تخمی، تنوع ژنتیکی زیاد میشود. یعنی برای یک ویژگی خاص بیش از یک الگوی ژنتیکی در جمعیت مشهود باشد، همانطور که میبینید مثال تنوع ژنتیکی با مثال انتخاب تکاملی با یکدیگر از جنبههایی متفاوت میباشند اما همانطور که میدانیم؛ تنوع ژنتیکی در یک جمعیت میتواند روی نرخ انتخاب طبیعی تأثیر داشته باشد.