- 668
- ۱۴۰۲/۰۲/۰۶ - ۰۶:۱۰
- 650 بازدید
شرح فصل و نکات ویژه: * در این فصل به بانکهای اطلاعاتی ای که هدف خود را معطوف به توالیهای اسیدآمینه کردهاند خواهیم پرداخت و همچنین بانکهایی معرفی میشوند که به ارائه ساختارهای سه بعدی پروتئینها میپردازند. * بانکها و ابزارهایی که معرفی میشوند را در اینترنت بازبینی کنید و با آنها کار کنید. * ساختار سوم بسیاری از پروتئینها که توسط روشهای آزمایشگاهی کشف شدهاند به صورت رایگان در بانکها قابل دریافت میباشد. * بحث در[…]
شرح فصل و نکات ویژه:
* در این فصل به بانکهای اطلاعاتی ای که هدف خود را معطوف به توالیهای اسیدآمینه کردهاند خواهیم پرداخت و همچنین بانکهایی معرفی میشوند که به ارائه ساختارهای سه بعدی پروتئینها میپردازند.
* بانکها و ابزارهایی که معرفی میشوند را در اینترنت بازبینی کنید و با آنها کار کنید.
* ساختار سوم بسیاری از پروتئینها که توسط روشهای آزمایشگاهی کشف شدهاند به صورت رایگان در بانکها قابل دریافت میباشد.
* بحث در رابطه با پیشگویی ساختار سوم پروتئینها در فصل دوازدهم انجام شده است.
۷۳-پایگاههای اطلاعات اولیه توالیهای اسیدآمینه و ساختار فضایی پروتئینها
در بخش اول این فصل تعدادی از پایگاههای توالیهای پروتئینی و روش جستوجو در آنها توضیح داده میشود. دادههای موجود در این پایگاهها عمدتا از ترجمه دستی یا خودکار توالیهای نوکلئوتیدی موجود در پایگاههای دیگر سرچشمه گرفته است.
۱-۴ پایگاههای دادهی توالی آمینو اسیدی
مارگارت دیهوف در بنیاد ملی پژوهش زیستپزشکی (NBRF) در واشینگتن، در جمعآوری توالیهای تمام آمینواسیدهای موجود فعالترین پژوهشگر بود و اولین اطلاعات را در اطلس توالیها و ساختارهای پروتئینی از ۱۹۶۸ تا ۱۹۷۸ منتشر کرد.
- مجموعه “اطلس توالیها و ساختارهای پروتئینی“:
- مفهوم ابرخانوادههای پروتئینی از این مجموعه پدید آمده و ماتریس دادههای جهش، معروف به MDMVA یا PAM براساس فراوانی جهشهای مشاهده شده در آمینواسیدها تدوین شد.
- در حدود سال ۱۹۸۰ یعنی زمانی که رقابت برای تاسیس یک پایگاه داده ملی DNA در آمریکا وجود داشت، این مجموعه محاسباتی رایانهای شد و پایگاه توالی پروتئینی NBRF نامگذاری شد.
- پایگاه دادهی منبع اطلاعات پروتئین Protein Information Resource (PIR) که در ۱۹۸۴ و با حمایت NIH تاسیس شد از پایگاه NBRF منشا گرفت.
- از ۱۹۸۸، PIR برای ایجاد پایگاه بینالمللی توالی پروتئین PIR با مرکز اطلاعات مونیخ برای توالیهای پروتئین
MIPS (Munich information Center for Protein Sequences) و پایگاه بینالمللی داده توالی ژاپن JIPID (Japanese International Protein Sequence Database.) همکاری میکند. - بنیاد پژوهش پروتئین PRF (Fundation Protein Research) در ازاکا ژاپن از سال ۱۹۷۵ تاکنون یک نشریهی چاپی Peptid information منتشر کرده است که شامل فهرستهای کتابشناسی از گزارشهای مربوط به پپتیدها و پروتئینهاست. این پایگاه داده رایانهای شده LITDB نام دارد.
- مدتها قبل از اینکه NCBI اتصالهای بانکهای مدلاین را معرفی کند، LITDB و SEQDB از ابتدا با یکدیگر ارتباط داشتند و از منابع مشترک استفاده میکردند.
- SEQDB غیر از دادههای مربوط به توالی اطلاعات دیگری ندارد و همچنان مجموعهی منحصر بهفردی است که حیطهی توالییابی پروتئین را پوشش میدهد.
- NBRF و PRF پایگاههای دادهی خود را با دیدگاهی متفاوت ایجاد کردند.
- NBRF: توالی آمینو اسیدی را یک هستی زیستشناختی میدانست. بخش اصلی کوشش NBRF بر حاشیهنویسی، ردهبندی ابرخانوادهها و حذف ورودیهای تکراری متمرکز کرد که متاسفانه سبب شد نتواند با سرعت افزایش دادهها همگام شود.
- PRF: توالی آمینو اسیدی را بخشی از اطلاعات کتابشناسی و مانند چکیدهی مقالات میدانست. این پایگاه در مقابل افزایش دادهها موفق بود اما کارایی پایگاه داده محدود بود.
- SWISS-PROT پایگاه داده توالی پروتئین دیگری بود که در سال ۱۹۸۶ در دانشگاه ژنو ایجاد شده اما خیلی زود از لحاظ کیفیت دادهها تبدیل به بهترین شد. داشتن ویژگیهایی همچون حاشیهنویسی قوی، تصحیح مداوم و دقیق دادهها و ارتباط با بسیاری از پایگاههای دادهی دیگر از خصوصیات بارز این پایگاه میباشد. Swiss-Prot با EMBL (آزمایشگاه زیستشناسی مولکولی اروپا) همکاری کرده است و ترجمهی توالی نوکلئوتیدی پایگاه داده EMBL یعنی (TREMBL) برای تکمیل Swiss-Prot استفاده شده است. در حال حاضر موسسه بیوانفورماتیک سوئیس (SIB) و موسسهی اروپایی بیوانفورماتیک EBI بهطور مشترک Swiss-Prot را اداره میکنند.
۱-۱-۴ جستوجو در بانک پروتئین NCBI:
در فصل گذشته با تعدادی از بانکهای پایگاه NCBI آشنا شدید ودر این فصل با دو بانک دیگر از این پایگاه آشنا میشویم به نام بانکهای Protein و Structure. در اینجا بانک Protein شرح داده میشود و در انتهای فصل به بانک structure خواهیم پرداخت.
۷۴-فصل چهارم
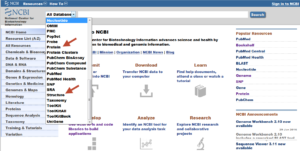
تصویر ۱-۴: نمایش لینک بانکهای Protein و Structure در منوی کشویی پایگاه NCBI.
توالیهای اسید آمینه ای در این بانک ذخیره میشوند. جستوجو در بانک پروتئین همانند بانک نوکلئوتید میباشد که در تصویر زیر مشاهده میکنید.ابتدا بانک protein را از منوی کشویی انتخاب کرده و در کادر روبهروی آن موضوع مورد جستوجو را نوشته و روی search کلیک کنید.
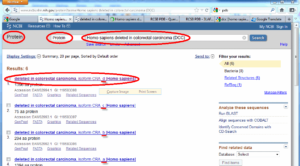
تصویر ۲-۴: نتیجه جستوجوی بانک protein
صفحه نتایج همانند صفحه نتایج بانک نوکلئوتید میباشد که در تصویر ۳-۴ میبینید که شامل سه بخش Header، Features و توالی میباشد.
۷۵-پایگاههای اطلاعات اولیه توالیهای اسیدآمینه و ساختار فضایی پروتئینها

تصویر ۳-۴: یک نمونه رکورد بانک Protein.
۲-۱-۴ پایگاه اطلاعات پروتئینی SwissProt
پایگاه توالی پروتئینی SwissProt در سال ۱۹۸۹ پایهگذاری شده است. این پایگاه حاوی اطلاعات توالی است که با ساختار ویژه به نمایش گذاشته میشود. یعنی هر فیلد در یک سطر قرار داده میشود. دو حرف اول هر سطر گویای نوع فیلد میباشد. درج اطلاعات به این شیوه اجازه تبادل آنها بین نرمافزارهای مختلف را میدهد.
SwissProt دارای سه ویژگی است:
- نامگذاری و تفسیر توالیهای پروتئینی موجود.
- کمترین تکرار (Non-redundancy).
- ارتباط منسجم (Integration) با بانکهای داده دیگر (با حداقل ۶۰ پایگاه اطلاعاتی ارتباط دارد).
۷۶-فصل چهارم
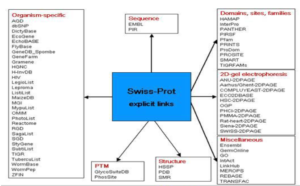
تصویر ۴-۴: نمایش ارتباط پایگاه توالی پروتئینی SwissProt با سایر بانکها.
در این پایگاه دو گروه داده وجود دارد:
- دادههای اصلی (Core data) شامل دادههای توالی، اطلاعات مربوط به citation، دادههای تاکسونومی.
- دادههای بدست آمده از نامگذاری و تفسیر توالیها شامل عملکرد پروتئین، تغییرات پس از ترجمه، دامینها و جایگاههای مهم در توالی، ساختار دوم، ساختار سوم، شباهت با پروتئینهای دیگر، بیماریهای مرتبط با پروتئین و واریانتهای آن.
برای جستوجو در این پایگاه وارد سایت http://expasy.org شوید و در این صفحه All databases را برای کلید واژه دلخواه مورد جستوجو قرار دهید.
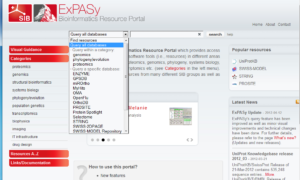
تصویر ۵-۴: تصویری از صفحه نخست پایگاه expasy و منوی کشویی آن که بانکهای فراوانی را در خود جای داده است.
۷۷-پایگاههای اطلاعات اولیه توالیهای اسیدآمینه و ساختار فضایی پروتئینها
در نتیجه جستوجو برای کسب اطلاعات در رابطه با پروتئین مورد نظر بر روی لینک uniprotKB کلیک میکنیم تا به این پایگاه منتقل شویم و سایر اطلاعات را در این پایگاه مشاهده خواهید کرد. لازم به ذکر است دادههای بانک Swiss prot در UniprotKB نمایش داده میشود. علت این که ابتدا از طریق پایگاه EXPASY جستوجو را شروع کردیم این بوده است که با پایگاه ExPASY آشنا شوید. این پایگاه شامل بانکهای زیادی میباشد که اکثراً در زمینه اطلاعات پروتئینی میباشند.
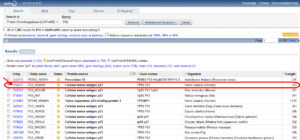
تصویر ۶-۴: لیست نتایج جستوجو در پایگاه uniprotKB .
از لیست موجود براساس نام پروتئین و ارگانیسم بر روی نام پروتئین مورد نظر کلیک میکنیم و اطلاعات در صفحهای نسبتا طولانی نمایش داده میشود که شامل موارد زیر میشود. یعنی برای هر پروتئین تمام اطلاعات سیزدهگانه زیر را نمایش میدهد و توسط لینکهای فراوان کاربران را به سایر بانکها راهنمایی میکند.
۱: Names
۲: Attributes
۴: Ontologies·
۵: Interactions
۶: Alt products
۸: Sequences
۹: References
۱۰: Web links
۱۱: Cross-refs
۱۲: Entry info
۱۳: Documents
معرفیUniProt :
منبع عمومی پروتئین (The Universal Protein Resource) جامع ترین کاتالوگ جهانی پروتئینهاست. این پایگاه محل ذخیره کلیه توالیهای پروتئینی و عملکرد آنهاست که از اشتراک دادههای SWissPort، TrEMBL و PIR به وجود آمده است. دادهها بهطور دستی (با دخالت کارکنان مربوط) نام گذاری میشود و دادهها در پایگاهی به نام UniPort Knowledgebase یا UniProtKB ذخیره میشوند. اگر نتیجه جستجوی شما حاصل یک ترجمه کامپیوتری از روی توالیهای DNA باشد با علامت یا متن مخصوصی این مورد را به شما تذکر خواهد داد. بخشی از این پایگاه تحت عنوان UniRef (The Uniprot Reference Clusters)
دادههای توالیهای پروتئین را براساس شباهت، خوشهبندی (Cluster) کرده است. همچنین بخشی دیگر تحت عنوان UniParc (The Uniprot Archive) در UniProt وجود دارد که دادههای غیرتکراری پروتئینها را ارائه میدهد.
پایگاه اطلاعات پروتئینی PIR(protein information resource):
۷۸-فصل چهارم
مخزن اطلاعات پروتئینی (The protein Information Resource) در بنیاد ملی تحقیقات بیوشیمی (NBRF) دانشگاه جورج تاون قرار دارد و مرکزیتی برای بانکهای اطلاعات پژوهشهای ژنومیک و پروتئومیک میباشد.
پایگاه دادههای زیر در جایگاه PIR وجود دارد:
iProClass
PIRSF
PIR-PSD
PIR-NRF
UniProt
۳-۱-۴ معرفی iProClass
این پایگاه اطلاعات ارزشمندی را برای uniProtKB و بخش پروتئینی NCBI فراهم میکند. در این پایگاه توالیهای غیرتکراری (non-redundan) پروتئینی ساماندهی شده و با بیش از ۹۰ پایگاه دادههای دیگر ارتباط متقابل دارد.
کاربردهای پایگاه iProClass به قرار زیر است:
- نام گذاری توالی پروتئین
- تهیه اطلاعات به روز با تفسیر برای هر پروتئین
- تهیه نقشه تشخیص هویت پروتئین (protein ID maping)
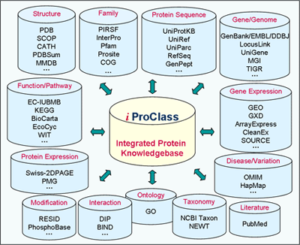
تصویر ۷-۴: نمایش ارتباط بانک iProClass با سایر بانکها و پایگاهها.
از منوی databases بر روی iProClass کلیک کنید .وارد صفحه زیر میشوید.
۷۹-پایگاههای اطلاعات اولیه توالیهای اسیدآمینه و ساختار فضایی پروتئینها

تصویر ۸-۴: نمایش لینک دسترسی به iProClass و سه بانک اصلی دیگر در پایگاهPIR.
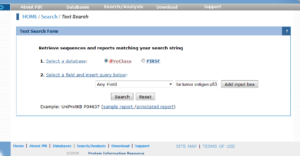
در سمت راست صفحه بر روی text search کلیک کنید.
تصویر ۹-۴: نمایش نحوههای مختلف جستوجو در iProClass .
در صفحهای که باز میشود پروتئین مورد نظر را جستوجو کنید.
۸۰-فصل چهارم
تصویر ۱۰-۴: نحوی جستوجوی Text در iProClass.
نتیجه به صورت زیر نمایش داده میشود.
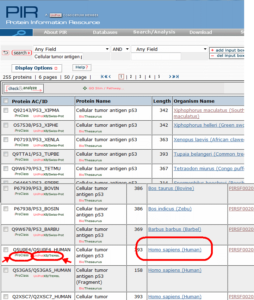
تصویر ۱۱-۴: لیست نتایج جستوجوی iProClass.
اگر بر روی uniport کلیک کنید به بانک uniport منتقل میشوید و اگر بر روی iProClass کلیک کنید اطلاعات مربوط به این بانک نمایش داده میشود که در تصویر ۱۲-۴ یک نمونه رکورد iProClass را مشاهده میکنید.
۸۱-پایگاههای اطلاعات اولیه توالیهای اسیدآمینه و ساختار فضایی پروتئینها
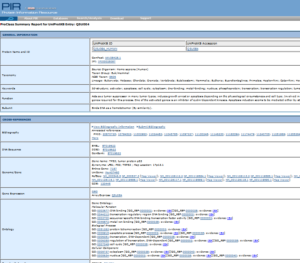
تصویر ۱۲-۴: یک نمونه رکورد iProClass.
۴-۱-۴ معرفی OWL
OWL یک بانک اطلاعات پروتئینی غیرتکرای است براساس SWISS-PROT به اضافهی توالیهای مربوط به
NBRF /PIR، GenPept و پایگاه ساختار سهبعدی PDB (NRL3D) است. OWL در دانشگاه لیدز (انگلستان) و با همکاری Bleasby از HGMRRC (مرکز مرجع پروژهی نقشهیابی ژنوم انسان) نگهداری میشود. متاسفانه این پایگاه تقریبا هر سه ماه یکبار گزارش میدهد و به صورت منظم بهروز رسانی نمیشود.
۲-۴ پایگاه دادههای ساختار فضایی پروتئینها
یکی از کارهای جالب بشری به تصویر کشیدن مفاهیم علمی است. تصاویر، مفاهیم علمی را گویاتر و جذابتر میکنند. زیستشناسان و محققین علم ژنتیک نیز از این ابزار به خوبی بهره برده و از آن در کارهای تحقیقاتی و انتقال مفاهیم به دانشاندوزان استفاده کردهاند. دستاوردهای زیادی در همکاری مهندسین کامپیوتر با دانشمندان زیستشناس حاصل شده است که این تلاشها در خور تقدیر است.
تجسم پیچو خمهای DNA و ماکرومولکولهای مرموزی چون پروتئینها و درک ویژگیهای وابسته به این ساختارها، برای انسان بسیار مشکل است. در این زمینه تصاویر سه بعدی بسیار راهگشا و پر فایده هستند. تصاویر و انیمیشنهای زیادی به همراه نرمافزارهای باارزشی که این تصاویر را نمایش دهند به وجود آمدهاند که در این فصل به نمونهای از آنها توجه خواهیم کرد. پژوهشگران به روشهای مختلفی از جمله تابش اشعه ایکس ساختار سه بعدی پروتئینها را مورد بررسی قرار میدهند. نتایج حاصل از این پرتونگاریها، اطلاعاتی است که در به تصویر کشیدن این ساختارها مورد نیاز است.
۸۲-فصل چهارم
۱-۲-۴ :(Protein Data Bank) PDB
در این پایگاه اطلاعات مربوط به ساختار سه بعدی ماکروملکولهای زیستی ذخیره شده است. در این پایگاه هم اکنون چند هزار ساختمان DNA، پروتئین یا هردو ذخیره شده است که همگی با روشهای آزمایشگاهی نظیر
NMR, X-ray diffraction, electron microscopy و مانند آن به دست آمدهاند.
بانک داده پروتئین PDB در سال ۱۹۷۱ در آزمایشگاه ملی بروکهَوِن [۱]BNL به عنوان پایگاهی، ساختارهای سهبعدی درشتمولکولهای زیستی که بهطور تجربی تعیین شدهاند ایجاد شد. در ۱۹۹۹ فعالیت PDB به [۲]RCSB منتقل شد. PDB همچنین حاوی دادههای ساختاری مربوط به پروتئینها، RNAها، قطعات کوتاه DNA، کربوهیدراتها، کمپلکسهای مولکولی و نیز ویروسها را دربرمیگیرد. PDB هر مدخل شامل مختصات اتمی، فهرستهای کتابشناسی، اطلاعات ساختار اولیه و ثانویه، همراه با فاکتورهای ساختاریابی بلورنگاری و دادههای تجربی NMR است. دادههای این پایگاه بهصورت روزافزون در حال افزایش میباشد که در تصویر ۱۳-۴ این رشد اطلاعاتی را مشاهده میکنید.
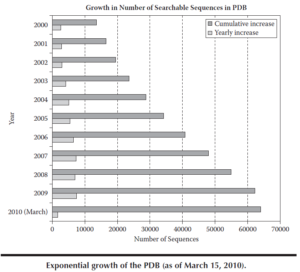
تصویر ۱۳-۴: نمایش نمودار رشد اطلاعات پایگاه PDB (March 2010)
برای ورود به این پایگاه به آدرس www.rcsb.orgمراجعه کنید.
[۱] Brookhaven National Laboratory
[۱] Research Collaboraory for Structural Bioinformatics
۸۳-پایگاههای اطلاعات اولیه توالیهای اسیدآمینه و ساختار فضایی پروتئینها
تصویر ۱۴-۴: صفحه اصلی پایگاه PDB.
هنگام جستوجو در PDB همچون سایر بانکهای اطلاعاتی میتوانید نام پروتئین مورد نظرتان را در باکس مخصوص جستوجو تایپ کنید. ما در اینجا پروتئین P53 را مورد جستوجو قرار دهیم.
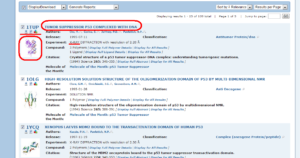
تصویر ۱۵-۴: لیست نتایج جستوجوی پایگاه PDB.
هر رکورد PDB شامل اطلاعات شناسنامهای (مانند عنوان، نام نویسندگان و …)، اطلاعات آزمایش انجام شده (نام روش، میزان تمایز و…) و بالاخره تصویر ثابت (still image) و ساختمان سه بعدی قابل دستورزی پروتئین (۳D-Structure) است.
۸۴-فصل چهارم
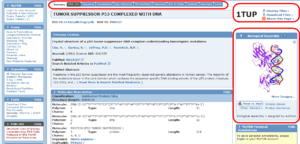
تصویر ۱۶-۴: یک نمونه رکورد PDB.
برای دستیابی به توالی اسید آمینه با فرمت FASTA و همچنین مشاهده ساختار دوم پروتئین میتوان وارد سربرگ Seqence شد.
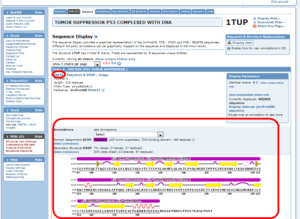
تصویر ۱۷-۴: نمایش ساختار دوم در رکوردهای PDB.
برای مشاهده ساختمان سه بعدی و چرخش آن نیاز به نرم افزارهای نمایشگر(PDB viewer) است. برخی از آنها مانند Chime MICE, Rasmol و VRML به طور مستقیم از همین پایگاه قابل دریافت است. از طریق ابزار Jmol که به صورت آنلاین در دسترس است، میتوان ساختار سوم را بهطور دقیق مشاهده کرد. برای مشاهده توسط Jmol شما نیاز دارید که JAVA بر روی سیستم شما نصب باشد.
۸۵-پایگاههای اطلاعات اولیه توالیهای اسیدآمینه و ساختار فضایی پروتئینها
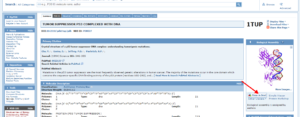
تصویر ۱۸-۴: دسترسی به نر افزار نمایش سه بعدی ساختار پروتئین به صورت آنلاین.
در صورتی که نرمافزار java بر روی کامپیوتر شما نصب باشد، ساختار سه بعدی پروتئین نمایش داده میشود که نمونهای از آن را در تصویر ۱۹-۴ میبینید.

تصویر ۱۹-۴: نمایش ساختار سوم پروتئین توسط Jmol.
۲-۲-۴ ساختار سه بعدی پروتئینها در NCBI
یک گروه از پایگاه دادهها به نام Structure توسط NCBI تدارک دیده شده است که اطلاعات مربوط به ساختار سه بعدی پروتئینها را در خود نگه میدارد. این گروه شامل موارد زیر است:
MMDB: پایگاه دادهای شامل ساختار سه بعدی ماکرومولکولها (The Molecular Modeling DataBase) به همراه ابزارهایی برای نمایش و مقایسه این ساختارها است. ساختار سه بعدی این پروتئینها به روشهای تجربی و از پروتئینهای موجود در پایگاه داده PDB (پایگاه داده پروتئینها) حاصل شده است. نرمافزاری به نام Cn3d توسط NCBI تدارک دیده شده است که به وسیله آن میتوان این ساختارهای سه بعدی را مشاهده کرد. شما میتوانید با توالی یک پروتئین شروع نمایید و با استفاده از BLAST تمام پروتئینهایی که با آن توالی شباهت دارند را مشخص نمایید و سپس ساختار سه بعدی هر یک را که تاکنون شناخته شده است را توسط ابزارهای نمایشگر مشاهده نمایید.
PubVats: پایگاه دادهای حاوی نتایج مقایسهStructure-Structure در مورد پروتئینها است. هر زنجیره پروتئینی به همراه دامین سه بعدی موجود در پایگاه MMDB با تمام پروتئینهای دیگر به وسیله الگوریتم VAST مقایسه میشود. لیستی از پروتئینهای مشابه حاصل از این مقایسه، از طریق لینکهای موجود در صفحات اینترنتی پایگاه داده MMDB بخش Summary موجود است که در این فصل در مورد آنها و نحوه استفاده از آن توضیح خواهیم داد.
PubChem: پایگاه دادهای حاوی اطلاعاتی در مورد فعالیتهای بیولوژی مولکولهای کوچک میباشد.
:CDD پایگاه داده دامینهای حفظ شده (Conserved Domain Database) میباشد.
آیا میتوان عملکرد یا ساختار پروتئینهای ناشناخته را پیشبینی کرد؟ آیا پروتئینهای ناشناخته عملکرد مشابه آنچه که در مورد پروتئینهای دیگر میدانیم دارند؟ آیا پروتئینهای با ساختارهای مشابه، عملکرد مشابهی دارند؟ و برعکس. اینها
۸۶-فصل چهارم
سوالهایی است که در ذهن بسیاری از محققین وجود دارد. نمایش سه بعدی پروتئینها از اهمیت خاصی برخوردار است. از این پایگاه داده میتوان جهت مقایسه پروتئینها با یکدیگر استفاده کرد. با مقایسه ساختار پروتئینها و یافتن الگوهای مشابه، میتوان در مورد عملکرد آنها پیشبینی کرد یا با عملکردهای مشابه پی به ساختارهای مشابه برد. اینجا با نحوه استفاده از این پایگاه دادهها آشنا میشوید.
پایگاه داده Structure
برای دسترسی به پایگاه داده Structure وارد سایت NCBI شوید و از طریق منوی کشویی وارد پایگاه داده Structure شوید. در جلوی عبارت Structure کادر سفیدی را مشاهده مینمایید که محل تایپ نام پروتئینی است که در نظر دارید آن را جستوجو نمایید. نمونهای را به عنوان جستوجو برای شما در نظر گرفتهایم تا روش استفاده از این پایگاه داده را فرابگیرید. ما هموگلوبین (hemoglobin) را به عنوان پروتئین مورد جستوجو انتخاب کردهایم و نام آن را در کادر اول تایپ کرده و بر روی کلمه Search کلیک نمودهایم تا صفحه زیر باز گردد.
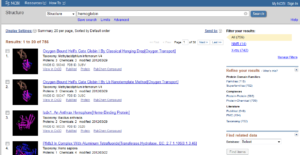
تصویر ۲۰-۴: نمایش لیست جستوجو در پایگاه Structure.
همچنان که در تصویر مشاهده مینمایید تعداد ۷۵۶ رکورد در مورد این پروتئین یافت شده است. در این صفحه تعداد ۲۰ رکورد از ۷۵۶ رکورد نمایش داده شده است. این ۲۰ رکورد با شماره از یکدیگر جدا شدهاند. در کنار هر شماره کدی را مشاهده مینمایید که کد این پروتئین در پایگاه داده PDB است. مثلا در مورد رکورد اول، کد این پروتئین ۳UBV است.
در انتهای هر رکورد کد این پروتئین در پایگاه داده MMDB است که مثلا در مورد رکورد اول به صورت
MMDB ID : 98348 نمایش داده شده است. اکنون آمادهایم تا اطلاعات بیشتری در مورد هر رکورد کسب نماییم. برای این منظور بر روی نام پروتئین که به صورت لینک و آبیرنگ است کلیک نمایید. به عنوان نمونه بر روی اولین رکورد که کد آن ۳UBV است، کلیک میکنیم که نتیجه را در تصویر بعد مشاهده خواهید کرد. این صفحه بهعلت طولانی بودن در دوتصویر تقسیم شده است.
۸۷-پایگاههای اطلاعات اولیه توالیهای اسیدآمینه و ساختار فضایی پروتئینها
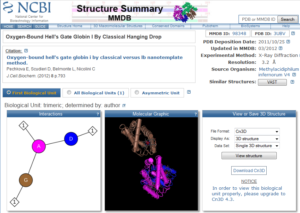
تصویر ۲۱-۴:یک نمونه رکورد پایگاه Structure. (قسمت اول)
عنوان پایگاه داده MMDB را در بالای صفحه مشاهده مینمایید. به این صفحه اینترنتی در مورد ساختار هر پروتئین، صفحه Summary گفته میشود که در زیر عنوان نام این پایگاه داده به صورت Summary مشاهده میکنید. در این صفحه خلاصهای از ساختار این پروتئین را میبینید که در ادامه جزییات هر بخش را شرح میدهیم:
Reference: لینک به مقالهای در پایگاه داده PubMed
Description: نام و توصیف خلاصهای از پروتئین، تاریخ ثبت اطلاعات این پروتئین درون PDB و لیست افرادی که ساختار پروتئین را تعیین کردهاند در این قسمت آمده است.
Source Organism: تاکسانومیهای مربوط به هر زنجیر پروتئین را لیست میکند.
MMDB ID: کد یکتای ساختار این پروتئین در پایگاه داده MMDB تنها با داشتن این کد میتوانید اختصاصا به اطلاعات و ساختار این پروتئین دست یابید. هرگاه پروتئین جدیدی وارد این پایگاه میشود، کد منحصر به فرد و جدیدی را دریافت میکند.
PDB ID: کد چهار حرفی این پروتئین در پایگاه داده پروتئینها. اگر بر روی این کد کلیک نمایید اطلاعات مربوط به این پروتئین را در پایگاه داده پروتئینها مشاهده خواهید کرد.
در قسمت بعد که با دکمهView 3D Structure مشخص شده است. میتوانید فایل مربوط به ساختار سه بعدی پروتئین را که در نمایش آن به وسیله نرمافزار Cn3D مورد نیاز است دانلود کنید.
جزییات بیشتر پروتئین در پایین صفحه اینترنتی به صورت گرافیکی نمایش داده شده است که به نام
Molecules and interactions مشخص شده است. در قسمتی که پروتئین را به صورت نوارهای قرمز و صورتی مشخص شده است روی Show annotation کلیک کنید با تصویر زیر مواجه میشوید.
۸۸-فصل چهارم
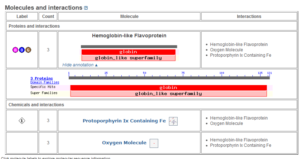
تصویر ۲۲-۴: بخش Molecules and interactions در یک رکورد پایگاه Structure. (قسمت دوم)
همانطور که در تصویر ۲۲-۴ مشاهده میکنید نوارها با جزئیات بیشتر مشخص شدهاند و یک خط کش به تصویر اضافه شده است و اگر بر روی این نوار درجهبندی شده کلیک نمایید شما را به پایگاه داده PubVast متصل میکند و در تصویر میبینید که تمام پروتئینهایی را که از نظر ساختاری شباهتی به هموگلوبین دارند را لیست کرده است.

تصویر ۲۳-۴: پایگاه داده PubVast.
۸۹-پایگاههای اطلاعات اولیه توالیهای اسیدآمینه و ساختار فضایی پروتئینها
در بالای تصویر ۲۳-۴ لوگوی پایگاه داده VAST را مشاهده مینمایید که عبارت simllar Structure در این قسمت اشاره به محتوای این پایگاه داده دارد. وظیفه این پایگاه یافتن پروتئینهای مشابه از نظر ساختاری با استفاده از الگوریتم VAST است. محققینی که بر روی ارتباط پروتئینها با یکدیگر از نظر ساختاری کار میکنند این صفحه برای آنها حایز اهمیت است.
در بالای این تصویر با عنوان VAST related structures for: MMDB ۹۸۳۴۸, ۳UBV sequence A. نشان داده شده است. یعنی این که در این صفحه، پروتئینهای مشابه (از نظر ساختاری) با این کد را لیست کرده است. در این قسمت از صفحه کنترلهای لازم برای مقایسه ساختار یا توالی پروتئینها با یکدیگر قرار داده شده است. برای استفاده از این کنترلها باید با مفاهیم و الگوریتمهای BLAST کردن آشنا باشید. در پایین صفحه پروتئینهای مشابه به صورت نوارهای قرمزی وجود دارند که اگر بر روی نام هر یک از پروتئینها کلیک نمایید صفحه ای باز میشود(تصویر ۷-۴) و زنجیرها و دیگر اطلاعات این پروتئین را نمایش میدهد.

تصویر ۲۴-۴:بخشی از رکورد پایگاه داده PubVast.
۳-۲-۴ CSD
CSD (پایگاه دادهی ساختاری کمبریج[۱]) شامل دادههای ساختار سهبعدی ترکیبات آلی و آلی – فلزی است که بعضی از آنها از لحاظ زیستشناختی اهمیت دارند. CSD بهوسیلهی مرکز دادههای بلورنگاری کمبریج (CCDC) که در سال ۱۹۶۵ در دانشگاه کمبریج آغاز به کار کرده و در سال ۱۹۸۹ یک موسسهی غیرانتفاعی شده، تهیه شده است. ساختارهای سهبعدی موجود در CSD توسط پراش اشعه x و پراش نوترون مشخص شدهاند. بانک CSD با سرعت ۱۵۰۰۰ ساختار در سال افزایش مییابد.
۳-۴ بانکهای اطلاعاتی خودکار
[۱] Cambridge Structural Database
۹۰-فصل چهارم
این بانکها بهوسیلهی رایانه تولید شده و حاوی ترجمهی توالیهای رمزکننده (CoDing Sequence = CDS) از یک بانک خاص حاوی دادههای DNA است:
TrEMBL: مجموعهای از CDSها از EMBL، به استثنای توالیهایی که قبلا در SWISS-PROT بودهاند، که در
EMBL-EBI با همکاری دانشگاه جنوا و موسسهی سوییسی بیوانفورماتیک نگهداری میشود. هر سه ماه گزارش داده و هفتگی بهروز رسانی میشود. دو قسمت در این بانک داده وجود دارد:
SP-TrEMBL: توالیهایی که برای وارد شدن به گزارش بعدی SWISS-PROT انتخاب شدهاند.
REM-TrEMBL: توالیهایی که وارد SWISS-PROT نمیشوند (مانند واریانتهای فرعی توالیهای یکسان، قطعات کوچک، توالیهایی مشکوک، توالیهای مصنوعی).
GenPept: مجموعهای از CDSها از GenBank، که در NCI-FCRDC (موسسهی ملی سرطان؛ مرکز توسعه و تحقیق سرطان فردریک؛ مریلند، آمریکا) نگهداری میشود. هر دو ماه گزارش میدهد و هر روز بهروز رسانی میشود.
تفاوتهایی در هدف و کیفیت میان این بانکهای داده وجود دارد برای نمونه SWISS-PROT بانک اطلاعاتی با مراقبتهای بالا است که حاوی مستندهای بسیار خوبی است. بهطور سیستماتیک واریانتها و قطعات را در یک ورودی (entry) منفرد ادغام میکند اما بعد از رشد بانکهای دادهای DNA، روند آن کند شده است. PIR حاوی توالیهای بیشتری است که شامل الیگوپپتیدهای توالییابی شدهی واقعی مختلفی است اما به خوبی کنترل نمیشود. بانکهای دادهای خودکار مانند TrEMBL و GenPept حتی بزرگتر هستند اما مستندهای کمی دارند و گاهی اوقات شامل ترجمههای خیالی هستند که بهطور واقعی در طبیعت وجود ندارند.