- 645
- ۱۴۰۲/۰۲/۰۶ - ۰۲:۵۶
- 592 بازدید
شرح فصل و نکات ویژه: * در این فصل با بانکهای اطلاعاتی اولیه که عموما حاوی دادههای آزمایشگاهی شامل توالیهای اسید نوکلئیک میباشند آشنا میشوید. * در این فصل به بانکهای حاوی توالی اسید نوکلئیک میپردازیم و در فصل بعدی به بانکهایی که دادههای اسیدآمینه را ارائه میدهند خواهیم پرداخت. * به بانکهایی که در این فصل معرفی شدهاند مراجعه کنید و در آنها جستوجو انجام دهید. ۵۳-پایگاههای اطلاعات اولیه توالیهای اسید نوکلئیک یکی از کاربردهای بیوانفورماتیک[…]
شرح فصل و نکات ویژه:
* در این فصل با بانکهای اطلاعاتی اولیه که عموما حاوی دادههای آزمایشگاهی شامل توالیهای اسید نوکلئیک میباشند آشنا میشوید.
* در این فصل به بانکهای حاوی توالی اسید نوکلئیک میپردازیم و در فصل بعدی به بانکهایی که دادههای اسیدآمینه را ارائه میدهند خواهیم پرداخت.
* به بانکهایی که در این فصل معرفی شدهاند مراجعه کنید و در آنها جستوجو انجام دهید.
۵۳-پایگاههای اطلاعات اولیه توالیهای اسید نوکلئیک
یکی از کاربردهای بیوانفورماتیک تحلیل دادهها جهت پیبردن به معمای تکامل هستی است. حل این معما در میلیاردها نوکلئوتید درون ژنوم موجودات زنده نهفته است. مشهورترین کاربرد بیوانفورماتیک در تحلیل توالیهاست. توالیهای DNA مربوط به ارگانیزمهای مختلف جهت دستیابی سریع و مقایسه آنها با یکدیگر، در پایگاههای داده ذخیره میشوند. پروژه ژنوم انسان که از سال ۱۹۹۰ تا سال ۲۰۰۳ به طول انجامید نمونهای از تحلیل توالیهاست. در این پروژه با استفاده از کامپیوترهای بزرگ و روشهای مختلف به دست آوردن توالیها، همه ژنوم انسان تعیین توالی گردید و درون یک پایگاه داده قرار گرفت. با کامل شدن نقشه ژنوم انسان، بیوانفورماتیک در تحقیقات سرطان به امید رسیدن به یک درمان موفق و نهایی بسیار با اهمیت شده است.
پایگاههای داده به دو دسته اصلی و فرعی تقسیم میشوند. نتایج تجربی حاصل از تحقیقات علمی مانند توالیهای نوکلئوتیدی یک ژن خاص، که در یک آزمایش تجربی به دست آمده است درون پایگاههای دادهی اصلی قرار میگیرند. این دادهها خام و بدون تحلیل هستند. نمونهای از این نوع پایگاهها GenBank است که توالیهای نوکلئوتیدی را نگهداری میکند. این پایگاه توسط NCBI مدیریت میشود. جدول ۲-۱ در فصل اول تعدادی از مهمترین پایگاههای بیوانفورماتیک را معرفی کرده است، یک ستون به نام Pri در این جدول پایگاههای اطلاعاتی اولیه را مشخص کرده است.
۱-۳ پایگاه دادهی توالی نوکلئوتیدی
در ۱۹۸۲ پایگاه دادهی GenBank در آزمایشگاه ملی لوسآلاموس و پایگاه داده EMBL در آزمایشگاه زیستمولکولی اروپا بهطوری رسمی کار خود را شروع کردند. بانک دادهی DNA ژاپن DDBJ در سال ۱۹۸۴ به آنها پیوست. در سال ۱۹۹۲ GenBank نقش خود را به NCBI واگذار کرد و فعالیت EMBL در ۱۹۹۴ به موسسه EBI منتقل شد.
– با آشکار شدن پروژههای ژنوم، پایگاههای داده نقش جدیدی برعهده گرفتند که کمک به کارهای آزمایشگاهی در حال اجرا بود.
– ساماندهی دادهها در پایگاههای داده ژنوم به جای اینکه کتابشناختی باشد، زیستشناختی است.
– درواقع پایگاههای داده ژنوم نشاندهندهی ساختارهای ژنوم با وضوح مختلف و کارکردهای ژنوم در سطوح مختلف است.
– نقشهای که بالاترین وضوح ساختار ژنوم را نشان میدهد، همان توالی نوکلئوتیدی ژنوم کامل است.
– در بسیاری از پایگاههای داده ژنوم، ژنها براساس سلسله مراتب عملکردیشان طبقهبندی شدهاند.
۲-۳ NCBI
در نوامبر ۱۹۸۸ بخش جدیدی به کتابخانه پزشکی ملی آمریکا (NLM) افزوده شد تا روشهای کامپیوتری پردازش اطلاعات جهت هدایت تحقیقات Biomedical ایجاد شود. این بخش مهم، مرکز ملی اطلاعات بیوتکنولوژی یا NCBI نام گرفت. کتابخانه ملی پزشکی آمریکا در سازمان ملی بهداشت آمریکا (NIH) قرار گرفته است. تجربههای موفق این کتابخانه در ایجاد و نگهداری پایگاههای داده در زمینه Biomedical و به عنوان بخشی از NIH باعث ایجاد یک برنامه تحقیقاتی در زمینه بیولوژی مولکولی کامپیوتری، شده است. امروزه NCBI به عنوان بزرگترین مرکز تحقیقات Biomedical در دنیا شناخته شده است. انستیتوی NCBI جهت رسیدن به اهداف خود فعالیت های زیر را دنبال میکند:
- هدایت تحقیقاتی در زمینه مسایل اصولی Biomedical در سطح مولکولی با بهرهگیری از روش های ریاضی و کامپیوتری.
- همکاری با انستیتوهای دیگر NIH، دانشگاهها، صنعت و دیگر سازمانهای دولتی.
- ایجاد ارتباط های علمی به وسیله برگزار کردن همایشها، کارگاههای آموزشی و سلسله سخنرانیها.
- برنامه های تحصیلاتی برای دانشجویان در زمینه های بیولوژی کامپیوتری.
- توسعه و انتشار نرم افزارها و پایگاههای داده مختلف.
سازمان NIH علاوهبر کتابخانه NLM از ۲۷ انستیتو و مرکز تحقیقاتی دیگر تشکیل شده است که از جمله این انستیتوها میتوان به موارد زیر اشاره کرد:
- انستیتوی ملی سرطان
- انستیتوی ملی تحقیقات ژنوم انسان
- انستیتوی ملی دیابت و بیماری های کلیه و گوارش
- انستیتوی ملی کودکان
۱-۲-۳ موتور جستوجوی Entrez
یکی از پر استفادهترین ابزارهای جستوجوی اطلاعات بیولوژی، موتور جستوجوی Entrez است که NCBI آن را ایجاد کرده است. این موتور جستوجو قادر است بهطور همزمان، بانکهای اطلاعاتی PubMed، توالیهای نوکلئوتیدی (GenBank)، توالیهای پروتئینی (protein)، ساختمان پروتئینها (Structure)، ژنوم کامل انسان و بعضی حیوانات، تاکسونومی، پایگاه داده بیماریهای ژنتیکی (OMIM) و بسیاری موارد دیگر را جستوجو کند.
۵۴-فصل سوم
موتور جستوجوی Entrez از ارتباط تنگاتنگ رکوردها در بانکهای مختلف استفاده میکند تا اطلاعات بیولوژی در مورد یک موضوع خاص را از پایگاه دادههای مختلف بازیابی کرده و در اختیار کاربر قرار دهد. بنابراین Entrez یک پایگاه داده نیست بلکه یک سیستم یکپارچه بازیابی اطلاعات است. محققین میتوانند از این موتور جستوجو بخواهند همه بانک های اطلاعاتی را همزمان جستوجو نماید یا این که جستوجوی خود را محدود به بانک اطلاعاتی خاصی نمایند. در این فصل نحوه استفاده از این موتور جستوجو را برای شما شرح میدهیم. جهت استفاده از این موتور جستوجو، وب سایت NCBI را با آدرس www.ncbi.nlm.nih.gov باز کنید. صفحه اصلی این سایت به صورت زیر است: (تصویر ۱-۳).
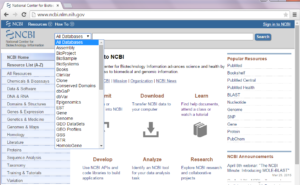
تصویر۱-۳: تصویری از پایگاه NCBI.
در بالا و سمت چپ این صفحه اینترنتی، لوگوی سایت NCBI را مشاهده می کنید. در سمت راست این لوگو دو جعبه وجود دارد که اولی مخصوص انتخاب پایگاه داده و دومی برای وارد کردن کلمات یا عبارت جستوجو است. بخش اول که به صورت یک منوی کشویی میباشد لیست پایگاههای اطلاعاتی NCBI را در اختیار شما قرار میدهد. گزینه All Databases به صورت پیش فرض برای شما انتخاب گردیده است تا همه بانکهای اطلاعاتی را جستوجو نماید. با کلیک بر روی فلش رو به پایین این منو آن را باز نمایید. بانکهای مهمی چون PubMed، Protein، Structrue و Books را در این منو مطابق تصویر ۱-۳ مشاهده مینمایید. در صورتی که بخواهید بانک خاصی را به تنهایی جستوجو نمایید بر روی نام آن بانک کلیک کنید.
برای جستوجوی همزمان در همه بانکها به وسیله موتور جستوجو Entrez با انتخاب گزینه All Database از منوی کشویی و تایپ موضوع جستوجو و کلیک بر روی گزینه Search به صفحه جدید وارد میشوید که در این صفحه نام پایگاههای دادهای که موتور جستوجوی Enterz بهصورت همزمان همه آنها را جستوجو کرده است، به چشم میخورد (تصویر ۲-۳). در کنار نام هر بانک عددی درج شده است که نشان دهنده موارد یافت شده از موضوع جستوجو در آن بانک میباشد.
۵۵-پایگاههای اطلاعات اولیه توالیهای اسید نوکلئیک
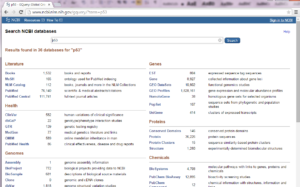
تصویر ۲-۳: نتیجه جستجوی موتور Entrez در تمام بانکهای پایگاه NCBI
۲-۲-۳ جستوجو در یک بانک اطلاعاتی خاص
علاوه بر جستوجوی همزمان همه بانکها میتوان یک بانک اطلاعاتی خاص را مورد جستوجو قرار داد. در صفحه اصلی NCBI از منوی کشویی بالای صفحه، بانک اطلاعاتی مورد نظر خود را انتخاب، و سپس عبارت جستوجوی خود را در کادر روبروی آن تایپ کنید و بر روی دکمه Search کلیک نمایید.برخی از مهمترین بانکهای اطلاعاتی که در NCBI در دسترس هستند را در این فصل به صورت کامل شرح خواهیم داد. و کمی در مورد تکنیکهای جستوجو صحبت خواهیم کرد. این تکنیکها به شما کمک خواهند کرد تا جستوجوهای موفقتری داشته باشید. لازم به ذکر است که این عبارات و تکنیکهای ارایه شده تنها در مورد این موتور جستوجو کاربرد دارد و در موتورهای جستوجوی عمومی مانند Google عمل نخواهد کرد.
۳-۲-۳ پایگاه توالی های نوکلئوتیدی:
توالیهای نوکلئوتیدی در سه پایگاه داده EMBL/DDBJ/Genbank نگهداری میشوند. دادههای جدید هر ۲۴ ساعت بین این سه پایگاه به اشتراک گذاشته میشود و دادهها را بین هم انتقال میدهند. همکاری بینالمللی پایگاههای توالیهای نوکلئوتیدی (INSDC) یک همکاری دیرین و بنیادین بین سه پایگاه داده ذکر شده در فوق میباشد.
۵۶-فصل سوم
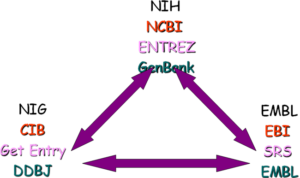
تصویر ۳-۳: ارتباط بین سه پایگاه داده EMBL/DDBJ/Genbank.
بنابراین دادههای هر سه بانک یکسان است و جهت امنیت و حفاظت از این دادهها، این سه بانک در سه گوشه دنیا قرار گرفتهاند. سازمان NCBI بانک نوکلئوتیدی بزرگ GenBank را مدیریت میکند. همچنان که گفته شد این سازمان در ایالت متحده قرار دارد. بانک نوکلئوتیدی DDBJ در ژاپن و EMBL نیز در اروپا استقرار یافتهاند. در این بخش چگونگی دستیابی به توالیهای نوکلئوتیدی موجود در پایگاه GenBank را شرح میدهیم. همانند بانکهای دیگر چندین راه برای دستیابی به این بانک نوکلئوتیدی وجود دارد. یک راه این که در صفحه اصلی NCBI جهت جستوجو، از منوی Search گزینه
All Database را انتخاب نمایید و جستوجویی را انجام دهید تا Entrez تمام بانکها را برای شما جستوجو نماید. سپس در صفحه نتیجه، لینک Nucleotide را انتخاب نمایید. راه سادهتر این که در صفحه اصلی NCBI منوی کشویی را باز و گزینهNucleotide را انتخاب نمایید در باکس روبهروی منوی کشویی چیزی تایپ نکنید و دکمه Search را کلیک نمایید تا صفحه اینترنتی مطابق تصویر۴-۳ با لوگوی Nucleotide را مشاهده کنید. در این صفحه توضیحاتی در رابطه با این بانک و همچنین لینکهای مفیدی برای دریافت اطلاعات بیشتر وجود دارد.
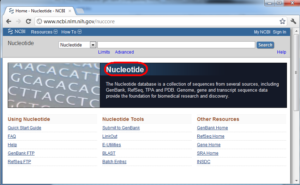
تصویر ۴-۳: صفحه اصلی پایگاه Nucleotide.
۱-۳-۲-۳ جستوجو در بانک نوکلئوتیدها
۵۷-پایگاههای اطلاعات اولیه توالیهای اسید نوکلئیک
به صفحه اینترنتی مطابق تصویر۴-۳ بروید و عبارت جستوجوی خود را در قسمت Search Box وارد و دکمه Search را کلیک کنید. هدف ما یافتن توالی نوکلئوتیدی ژن DCC میباشد. بعد از جستوجو نتیجه را در تصویر ۵-۳ مشاهده مینمایید. همانطور که ملاحظه میکنید تعداد ۴۴۰۸ رکورد یافت شده است که با شماره از هم جدا شدهاند. و در هر صفحه تعداد ۲۰ نتیجه جستوجو نمایش داده میشود.
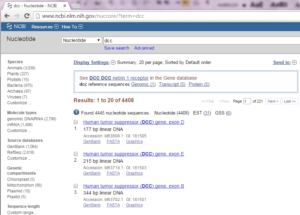
تصویر۵-۳:جستجو در پایگاه Nucleotide.
درباره هر رکورد اطلاعات زیر وجود دارد:
در سطر دوم در کنار هر شماره عبارتی به نام Accession را میبینید که با حروفی مثل NM، NR و … شروع شده است. این عبارت کد یکتای رکورد است که به آن Accession Number گفته میشود. در مراجعات بعدی تنها با این کد میتوانید به این رکورد دست یابید بدین ترتیب که این کد را به عنوان عبارت جستوجوی خود در Search Box وارد نمایید. بنابراین اگر در جستوجوها، رکوردی را مناسب تحقیقات خود دانستید کافی است که این شماره را یادداشت نمایید. برای درکAccession Numberها به سه جدول زیر که کدهای مربوط به GenBank را شرح داده اند توجه کنید.
جدول ۱-۳: Accession Numberها در GenBank.
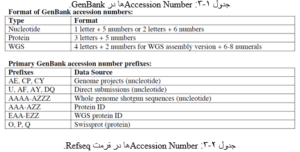
جدول ۲-۳: Accession Numberها در فرمت Refseq.
۵۸-فصل سوم
در سطر دوم عدد دیگری را بعد از GI میبینید که کد این رکورد در بانک GenBank است. اگر بر روی لینک هر رکورد کلیک کنید جزییات هر رکورد همراه با توالی نوکلئوتیدی آن مطابق تصویر۶-۳ نمایش داده میشود که ما در ادامه محتویات این رکورد را کاملا شرح میدهیم.
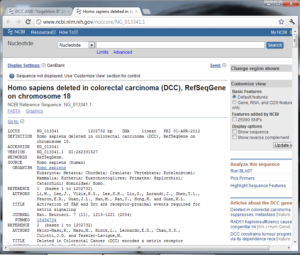
تصویر ۶-۳: یک نمونه رکورد بانک Nucleotide
۲-۳-۲-۳ بررسی یک رکورد نمونه
محتوای این صفحه (تصویر ۶-۳) بسیار طولانی است، برای شرح جزییات این صفحه، نمونهای کوچکتر را ارایه کردهایم و در پایان این رکورد نیز توضیحات کاملی درباره آن آمده است. در نگاه اول، این اطلاعات بغرنج و نامفهوم به نظر میرسند. جزء جزء این اطلاعات را مورد بررسی قرار داده و مفهوم آنها را شرح میدهیم. به این ساختار ارایه شده falt file گفته میشود. اطلاعات درون falt fileها به صورت Line type ارایه میشود. بدین معنا که در اول هر سطر، یک کلمه یا یک کد قرار گرفته است و بیان میکند در آن سطر چه اطلاعاتی گنجانده شده است. برای مثال در سطر اول کلمه LOCUS در ابتدای سطر، بیان می کند که در این سطر کد یکتای رکورد (SCU49845) یا ID Locus قرار دارد. یا در سطری که در ابتدای آن کلمه ORGANISM آمده، بیان کننده درخت تاکسونومی است که این ژن از آن گرفته شده است.
۵۹-پایگاههای اطلاعات اولیه توالیهای اسید نوکلئیک
نظر میرسند. جزء جزء این اطلاعات را مورد بررسی قرار داده و مفهوم آنها را شرح میدهیم. به این ساختار ارایه شده falt file گفته میشود. اطلاعات درون falt fileها به صورت Line type ارایه میشود. بدین معنا که در اول هر سطر، یک کلمه یا یک کد قرار گرفته است و بیان میکند در آن سطر چه اطلاعاتی گنجانده شده است. برای مثال در سطر اول کلمه LOCUS در ابتدای سطر، بیان می کند که در این سطر کد یکتای رکورد (SCU49845) یا ID Locus قرار دارد. یا در سطری که در ابتدای آن کلمه ORGANISM آمده، بیان کننده درخت تاکسونومی است که این ژن از آن گرفته شده است.
کالبدشکافی Flat Fileهای توالیهای نوکلئوتیدی
file Flatها به سه بخش اصلی تقسیم میشوند:
۱- Header حاوی اطلاعاتی است در مورد کل رکورد.
۲- Features (ویژگیها) که یادداشتها و توضیحات رکورد است.
۳- توالی نوکلئوتیدی.
همه file flatهای پایگاههای نوکلئوتیدی دارای علامت // در انتهای هر رکورد هستند. اکنون هر سه بخش بالا را مفصل شرح میدهیم.

۶۰-فصل سوم
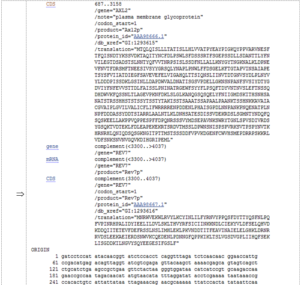
The header
بخش header بخشی است که بیش ترین وابستگی را به پایگاه داده دارد. یعنی این که در پایگاه دادههای نوکلئوتیدی مختلف تفاوتهایی مشاهده میشود. سطر اول همه flat fileها در بانک GenBank سطر LOCUS است که در زیر نمونهای از آن را میبینید:
LOCUS SCU49845 ۵۰۲۸ bp DNA PLN 23 MAR ۲۰۱۰
این المان (نام) باید با یک حرف شروع شود و کاراکتر بعدی میتواند حرف یا عدد باشد. در این مثال سه کاراکتر اول، حرف و بقیه عدد است. همه حروف نام همگی حرف بزرگ هستند. طول این المان باید از ۱۰ کاراکتر بیشتر نشود. در گذشته افرادی که توالیهای درون بانک را کنترل می کردند، میکوشیدند تا نام مفید و با معنی انتخاب کنند در ضمن این که این نام میبایست در کل بانک یکتا باشد، از آن جا که تمام نامهای با معنا تاکنون استفاده شده است، نامهای جدید معنای خاصی ندارند. امروزه این نام، تنها از این نظر در بانک حضور دارند که نرم افزارهای قدیمی زیادی وابسته به این فیلد هستند. بنابراین این نامها حاوی اطلاعات مفیدی نیستند.
دومین المان در خط LOCUS، طول توالی است. در این مثال طول توالی ۵۰۲۸ bp است.
سومین المان در این سطر نشان دهنده ماهیت بیولوژی مولکول است که معمولا DNA یا RNA است.
المان چهارم در این سطر (PLN)، کد تقسیمبندی (division code) است. این کد سه حرفی، کد تقسیمبندی تاکسانومی بوده یا برای دستهبندیهای دیگری به کار میرفته است، NCBI کد تقسیمبندی تاکسانومی ارگانیزمها را به روز نکرده است، چرا که فکر می کرد این کد ۳ حرفی مناسب نمایش گونههای متنوع بیولوژی موجود روی کره زمین نیست. این نوع کدگذاری امروزه منسوخ شده است. جدیدا این تقسیمبندی بر پایه عملکرد (functuion) ژنها صورت میگیرد. ممکن است به جای این کدها با عباراتی نظیر EST یا STS برخورد نمایید. توضیح مختصری از این دو کد در زیر آورده شده است:
۶۱-پایگاههای اطلاعات اولیه توالیهای اسید نوکلئیک
EST
این کلمه مخفف Expressed Sequence Tag است. این تقسیمبندی در سال۱۹۹۳ معرفی شده است. رکوردهایی که دارای این کد سه حرفی هستند، حاوی توالیهای کوتاه از cDNA هستند. معمولا به تعداد زیادی از روی این نوع ژن رونویسی صورت میگیرد. ESTها نمایش یک تصویر فوری از آنچه در یک بافت خاص یا در یک مرحله خاصی از رشد موجود (development) بیان میشود. اینها tagهایی از بین یک ژن خاص موجود در یک کتابخانه خاص cDNA هستند. (در فصل ترنسکریپتومیکسESTها به تفصیل شرح داده شدهاند)
STS
این کلمه مخفف “Sequence Tagged Site” است. STSها توالیهای کوتاه (بین ۲۰۰ الی ۵۰۰ جفت باز) و روی ژنوم یکتا هستند. جایگاه و توالی آنها کاملا مشخص است. این توالیها قابل آشکار شدن به وسیله PCR هستند. توالیهای STS، به دلیل یکتا بودن روی ژنوم، از آنها جهت تهیه نقشههای ژنتیکی استفاده میشود.
المان پنجم در خط اول تاریخ اضافه شدن این رکورد به GenBank است. در این مثال: ۲۳-MAR-2010. اگر هر یک از ویژگیهای رکورد به روز شده باشد و سپس رکورد انتشار یابد، این تاریخ، آخرین تاریخ انتشار است.
بخش بعدی header، سطر definition به فرمت زیر است:
DEFINITION Saccharomyces cereviiae TCP1-beta gane , partial cds, and Ax12p(AXL2) and Rev7p (REV7) genes, completed cds.
در این سطر، اطلاعاتی ارایه میشود که نشان دهد این رکورد راجع به چه موضوعی است. در ایجاد این خطوط دقت فراوانی صورت گرفته است. گرچه بسیاری از این اطلاعات را می توان از بخش های دیگر رکورد به دست آورد ولی این اطلاعات چنان انتخاب شدهاند تا دارای ارزش باشند. بنابراین در مراجعه به این رکورد سری به این سطر بزنید تا مطمئن شوید که اطلاعات رکورد مناسب تحقیقات شما است یا خیر. به هر حال همیشه امکان ندارد که تمام اطلاعات بیولوژیک رکورد را بتوان در یک سطر جمعآوری کرد. کاربران باید تنها به این سطر اکتفا نکنند.
کمی پایینتر، کد دسترسی (ACCESSION)، کلید اصلی برای ارجاع به رکورد در پایگاه داده را وجود دارد. کد دسترسی همیشه با رکورد خواهد بود و اگر رکورد تغییر کند (مثلا با تغییر یک نوکلئوتید) کد دسترسی تغیر نخواهد کرد. هم اکنون کد دسترسی به یکی از شکلهای روبهرو است: “۵+۱” و “۶+۲”
- “۵ +۱” یعنی یک حرف بزرگ انگلیسی در اول و پنج رقم به دنبال آن، در مثال بالا کد دسترسی U49845 است که از این قانون پیروی میکند.
- “۶+۲” یعنی دو حرف بزرگ انگلیسی در ابتدا و شش رقم به دنبال آن، اکثر رکوردهای جدیدی که امروزه وارد بانک میشوند کدی به صورت این فرمت دوم دارند.
سطر بعدی در این رکورد به VERSION تعلق دارد:
VERSION U49845.1 GI: 1293613
قالب version به صورت accession.version”” است (U49845.1). شماره accession ثابت بوده ولی version هر گاه که توالی تغییر کند یک واحد به آن افزوده میشود. سطر VERSION در GenBank همچنین دارای عدد
GI (the geninfo identifier) است. که در این مثال GI: 1293613 است. اگر توالی تغییر کند GI به عدد صحیح موجود بعدی تغییر می کند. عدد GI فقط مخصوص GenBank است. عدد accession.version به عنوان نام اصلی رکورد (identifier) است که تنها و تنها به یک رکورد در هر بانک اشاره می کند.
سطر بعدی حاوی کلمات کلیدی (keywords) رکورد است. این خطوط به دلایل تاریخی وجود دارند و در بسیاری از موارد متاسفانه بیاستفادهاند.
اطلاعات Taxonomy در سطرهایی با عنوان SOURCE و ORGANISM در GenBank وجود دارد. این کلاسبندی از بالا به پایین است. مطابق درخت taxonomic عمومیترین و بزرگترین گروه، اول آورده میشود.
هر رکورد GenBank باید حداقل یک مرجع (reference یا citation) داشته باشد. این مرجعها مقالاتی هستند که به توالی اعتبار می دهند که چرا این ژن خاص تعیین توالی شده است. در بسیاری از موارد رکوردها دو یا چند مرجع دارند.
اگر به رکورد اصلی مراجعه نمایید متوجه میشوید که سه مرجع با ساختار بالا وجود دارد. مرجعها با شمارهای که دقیقا جلوی کلمه REFERENCEقرار گرفته است از هم جدا شدهاند. افزودن یک رکورد به بانک برای فرد اعتبار علمی ایجاد میکند. هر مرجع از بخشهای زیر تشکیل شده است.
نویسندگان مقاله (AUTHORS)
عنوان مقاله (TITLE)
نام ژورنال و سال انتشار آن (JOURNAL)
کد PMID این مقاله (PUBMED)
۶۲-فصل سوم
از طریق این مراجع بین دو بانک نوکلئوتیدها و بانک PubMed ارتباط برقرار میشود. بدین ترتیب میتوانید با استفاده از PMID این مرجعها به مقالات مرتبط با این رکورد دست یابید.
The Feature Table
بخش میانی رکورد، FEATURES نام دارد. در قسمت FEATURES، اطلاعات ژنتیکی توالی قرار دارد. اطلاعات موجود در این قسمت دستهبندی شده است. دستهای از این اطلاعات در قسمت source قرار دارد که به کل ژن اشاره دارد و دستهای دیگر در قسمت CDS و بخشی نیز در قسمت gene قرار دارد.
تفسیر این اطلاعات به صورت زیر است:
طول این ژن ۵۰۲۸ نوکلئوتید است که در جلوی source به صورت ۱…۵۰۲۸ نشان داده شده است.
این ژن مربوط به ” ساکارومایسس سروسیه” است که در سطر /organism در بخش source قرار گرفته است.
کد تاکسانومی این موجود در سطر db_xref، عدد ۴۹۳۲ را نشان می دهد.
این ژن روی کروموزوم IX این موجود قرار دارد که در بخش /chromosome در زیر source مشاهده می کنید.
توالی این ژن از سه exon تشکیل شده است که اطلاعات هر کدام در یک CDS جداگانه گذاشته شده است. همچنان که در قسمت اولین CDS می بینید اولین اگزون از باز ۱ شروع و به باز ۲۰۶ ختم میشود. کدون شروع آن باز شماره ۳ است (codon_start=3/). نام این پروتئین TCP1-beta، شناسه آن AAA98665 در سطرهای بعدی آمده است. ترجمه این exon نیز در سطر translation مشاهده می نمایید این ترجمه توسط نرمافزار انجام شده است نام این ژن AXL2، در سطر gene قرار دارد.
توالی ژنها
در آخر رکورد نیز در بخش ORIGIN توالی کامل این ژن وجود دارد. در بسیاری از نرمافزارهای کامپیوتری از این توالی نوکلئوتیدی به عنوان ورودی نرم افزار استفاده میشود مانند نرم افزار BLAST که توالیهای نوکلئوتیدی را با یکدیگر مقایسه مینماید.
۴-۲-۳ معرفی GenBank
GenBank مشهورترین پایگاه دادههای توالی نوکلئوتیدی NCBI و مستندات مربوط است که به عنوان بخشی از کتابخانه ملی پزشکی (National Library of Medicine) در سال ۱۹۸۲ پایهگذاری شد. ابزارهای موجود برای دسترسی به دادههای این پایگاه عبارتند از BLAST (1990)، Entrez (1992)، GenBank (1992) و PubMed (1997). به دلیل تسلیم انواع دادههای ژنومی، رشد اطلاعات در این بانک بسیار سریع بوده است. بهطور میانگین، ماهانه سه میلیون توالی و ۱۴۰۰ گونه جدید به این بانک اطلاعاتی افزوده میگردد به طوری که تقریبا هر ۱۰ ماه حجم اطلاعات آن دو برابر میشود.
همان طور که پیشتر گفته شد، دادههای این پایگاه با بانک دادههای DNA ژاپن (DDBJ) و موسسه زیستشناسی مولکولی اروپا (EMBL) در حال تبادل بوده و هر سه پایگاه، اطلاعات خود را روزانه رد و بدل میکنند. GenBank حاوی دادههای تکراری و اضافی (مانند توالیهای وکتوری) است که بخشی از آن به دلیل تسهیل در ورود اطلاعات بوده است. اما دلیل عمده آن امکان تسلیم توالیهای مشابه تکراری از سوی آزمایشگاههای مختلف بوده است تا بتوان ذخیرهای از گوناگونی ژنتیکی (polymorphism) ایجاد نمود.
بخشهای عمومی GenBank که به بخشهای تاکسونومیک مشهورند، بر حسب نوع موجود عبارتند از:
BCT (Bacterial and Archea)
MAM (Mammalian)
Inv (Invertebrate)
PHG (Phage)
PLN (Plant and fungi)
PRI (Primate)
ROD (Rodent)
SYN (synthetic= cloning vectors)
VRL (Viral)
VRT (other vertebrate)
GenBank همچنین به دلایل فنی (ماهیت حجیم و با کیفیت پایین دادهها) بخشی از اطلاعات خود را در قسمتهای اختصاصی اطلاعات حجیم ذخیره میکند.
PAT (Patents)
۶۳-پایگاههای اطلاعات اولیه توالیهای اسید نوکلئیک
EST (Expressed Sequence Tag)
STS (Sequence Tagged Site)
GSS (Genome Survey Sequence)
HTG (High Throghput Genome)
CON (Contig)
بانک ژن با آدرس www.ncbi.nlm.nih.gov/genbank شناخته میشود اما در این آدرس امکان جستجو وجود ندارد و فقط اطلاعاتی در مورد بانک و شیوه ارائه داده ها به بانک شرح داده شده است. بانک ژن برای فراهم کردن و تقویت دسترسی جامعه علمی به اطلاعات جامع و به روز رسانی شده توالیها طراحی شده است.
روشهای تسلیم داده به بانکهای اطلاعاتی اولیه و توالی نوکلئوتیدی
دادههای موجود در GenBank و بانکهای مشابه از دو طریق تامین میشود:
۱:از تحقیقات پژوهشگران در دنیا
۲:از مراکز توالییابی ژنومها در دنیا به شکلهای مختلف EST، STS، GSS، HTC و توالیهای بلند موجود در کاسمیدها و BAC کلونها.
دادههای کم حجم و معمولا مشتمل بر یک توالی با استفاده از نرم افزار Bankit و دادههای ژنومی حجیم با استفاده از نرم افزار Sequin تسلیم میشوند. تسلیم اطلاعات از طریق On-line، email و یا حتی ارسال بسته اطلاعاتی مانند، فلاپی، سی دی، دی وی دی و غیره امکانپذیر است. بدیهی است که دادهها اکثرا از طریق WWW تسلیم میشوند. تسلیم توالیهای کوتاه cDNA مانند نتایج آزمایشهای نمایش بیان ژنها (Differential display) و یا ESTها از طریق بانک ESTها و روال مربوط انجام میشود. از اول ژانویه ۲۰۰۰، توالیهای غیر ژنومی بلند GSS از طریق Bankit پذیرفته نمیشوند و باید در dbGSS فرستاده شوند.
پایگاه دادههای Refseq
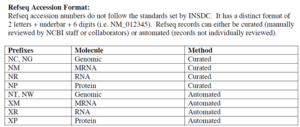
Refseq به مجموعه توالیهای مرجع استخراج شده از GenBank را گویند که تصحیح شده و غیر تکراری هستند. یعنی بر خلاف GenBank، در پایگاه Refseq هر رکورد مربوط به یک ژن یا فرم پیرایش شده از یک ژن میباشد. در بانک دادههای Refqseq شماره دسترسی هر مولکول با دستوری ویژه تعیین میشود که در جدول زیر مشخص شده است.

۶۴-فصل سوم
۵-۲-۳ تفاوت سه بانک Gene، Nucleotide، Genome
تصویر ۷-۳: نمایش لینک برخی از بانکها در منوی کشویی پایگاه NCBI.
شاید در نگاه اول گیجکننده باشد و نتوانیم راحت بفهمیم توالی مورد نظرمان را در کدام بانک جستجو کنیم اما با توضیحاتی که در ادامه آمدهاند مسئله روشن میشود.
بانک Gene
این بانک داده اطلاعات مربوط به یک ژن خاص(نه اطلاعات مربوط به کل ژنوم) را در اختیار کاربر قرار میدهد. این بانک اطلاعات مربوط به همه ژنها شناخته شده را ندارد و روی ژنومهایی که به طور کامل توالییابی شدهاند متمرکز میباشد. Entrez Gene بخشی در NCBI میباشد که اطلاعات مفیدی در مورد نقشه، توالی،بیان ژن،ساختار،عملکرد و هومولوژی را در اختیار همگان قرار میدهد. اطلاعات فراوانی از طیف گستردهای از گونهها موجود میباشد که سابقه نامگذاری، نقشهها، مسیرها، تغییرات، و لینکهایی به ژنوم، فنوتیپ، و لوکوس به بانکهای دیگر را دارا میباشد. همچنین این بانک حاوی دادههای RefSeqs میباشد. دامنه وسیعی از اطلاعات دربارهی ژنها و موجودات را در بر دارد. این اطلاعات شامل نتایج تجزیه و تحلیلهایی است که روی دادههای توالی صورت گرفته است. مقدار و نوع اطلاعات ارائه شده وابسته به این است که چه اطلاعاتی راجع به یک ژن و یا موجود مشخص در دسترس است و میتواند شامل ۱) خلاصه گرافیکی محتوی ژنومی، ساختار اگزون/ اینترون، ۲) تصویر گرافیکی توالی mRNA ، ۳) انتولوژی ژنی و اطلاعات مربوط به فنوتیپ، ۴) دادههای توالی پروتئینی و دمینهای حفظ شده ، ۵) پایگاههای اطلاعات مربوط به جهش میباشد.
بانک Genome
این پایگاه داده امکان دستیابی به اطلاعات ژنومی بیش از هزار گونه که به طور کامل توالی یابی و نقشهیابی شدهاند و یا این که توالییابی آنها در حال انجام است را فراهم میآورد. نمایش گرافیکی نتایج اطلاعات مفیدی را در سه سطح، ژنوم، یک کروموزوم و یک ژن در اختیار پژوهشگران قرار میدهد.
بانک Nucleotide
همانطور که پیشتر توضیح داده شد Gen bank پایگاه داده توالیهای نوکلئوتیدی ارگانیسمهای مختلف است و قابلیت جستجو در این بانک از طریق بخش Nucleotide فراهم میباشد. این مجموعه شامل توالیهای نوکلئوتیدی شامل mRNA، قطعات DNA ژنومی، ژنهای منفرد، ژنهای چند گانه و ژنهای دستهای rRNA میباشد که در دسترس عموم قرار دارد. پایگاه دادههای Nucleotide مجموعهای از منابع مختلف، از جمله GenBank، RefSeq، TPA و PDB است. این بانک حاوی دادههای بسیار گستردهای میباشد و اغلب جستجو در آن بسیار گیج کننده میباشد. معمولا محققین ابتدا از بانک Gene استفاده میکنند و در صورتی که نتوانند اطلاعات مناسبی پیدا کنند از این بانک استفاده میکنند.
۶۵-پایگاههای اطلاعات اولیه توالیهای اسید نوکلئیک
۶-۲-۳ بانکهای اطلاعاتی توالیهای غیرتکراری
هیچکدام از بانکهای اطلاعاتی موجود واقعا کامل نیستند. برای این کار تلاشهایی برای ایجاد بانکهای اطلاعاتی مرکب صورت گرفت. با مقایسهی توالیها و با حذف توالیهای تکراری و توالیهایی که بهطور جزیی با هم متفاوتاند، از تکراری بودن آنها ممانعت میشود. معیار دقیق برای تکراری بودن، این پایگاههای مرکب را نسبتا کوچک میکند و بنابراین در جستجوی بانکهای اطلاعاتی کارایی بهتری دارد.
۷-۲-۳ بانکهای اطلاعاتی حاوی توالیهای اختصاصی
علاوه بر بانکهای اطلاعاتی عمومی، چندین بانک اطلاعاتی اختصاصی وجود دارد. برخی از این بانکها از پایگاههای عمومی منشا گرفتهاند، بهطوری که برخی از آنها عضویت مولفان را پذیرفته و میتواننند حاوی توالیهایی باشند که هنوز در پایگاههای عمومی یافت نمیشود. آنها یک یا چند مزیت زیر را دارند:
* بانک اطلاعاتی تشکیل یک مجموعهی بسیار مشخص از توالیها را میدهد که پیدا کردن آنها در پایگاههای عمومی مشکل است. جستجوی پایگاه اختصاصی به جای پایگاه عمومی لیستی از توالیهایی را تهیه میکند که ناخالصی و توالیهای مزاحم و ناخواسته را کم میکند و در زمان کمتری مورد استفاده قرار میگیرد.
بانک اطلاعات اختصاصی شده معمولا غیرتکراری است و حاوی توالیهای یکسان نیست.
* گاهی تعریف موضوع دادهها یا کلمات کلیدی بهتر استانداردسازی میشود که به کاربر امکان یافتن توالیهای مورد نظر را با یک جستجوی تککلمهای میدهد. در یک بانک اطلاعاتی بدون استانداردسازی، جستجو باید با کلمات کلیدی متفاوت تکرار شود.
* مستندات نیز بیشتر و بهتر از بانک اطلاعات عمومی است.
چند مثال از بانکهای اطلاعاتی اختصاصی به شرح زیر است:
پایگاه اطلاعات HIV: توالیهای DNAی HIV و SIV که در LANL (آزمایشگاه ملی لوسآنجلس؛ نیومکزیکو، آمریکا) نگهداری میشود و سالیانه گزارش میدهد.
HPVSD: توالیهای پروتئینی و DNAیی از papillomavirusهای انسانی و حیوانی و پروتئینهای سلولی که به وسیلهی آلودگی این ویروسها تحت تاثیر قرار میگیرند؛ این پایگاه اطلاعاتی در LANL نگهداری میشود و سالیانه گزارش میدهد.
IMGT (ایمنوژنتیک): مجموعهای از بانکهای اطلاعاتی در زمینهی ایمنیشناسی.
IMGT /LIGM (آزمایشگاه ایمنوژنتیک مولکولی): ژنهای ایمنوگلوبولینها و گیرندههای سلولهای T که از EMBL گرفته شده و مستندات آن بهبود یافته و در دانشگاه مونتپولیر (فرانسه) نگهداری میشود.
IMGT /HLA (سازگاری بافتی جایگاه ژنی A در انسان): ژنهایی برای جایگاههای اصلی سازگاری بافتی در انسان که در موسسهی تحقیقاتی Anthony Nolan (لندن) نگهداری میشود.
NRL-3D: زیرمجموعهای از PIR، حاوی توالیهای پروتئینی برای آن پروتئینهایی که ساختار سهبعدی شناخته شده دارند و در PDB ثبت شدهاند، که در NBRF نگهداری شده و هر سه ماه گزارش میدهد.
۸-۲-۳ دیتابیسهای NCBI
با توجه به تعداد زیاد بانکهای موجود در سایت NCBI و اهمیت بالای آنها اکثر بانکهای این سایت با شرحی مختصر که برگرفته از کتاب راهنمای NCBI میباشد در ادامه آمده است. بسیاری از این بانکها در بخشهای مختلف این کتاب شرح داده شدهاند.
Mitelman
در این بانک داده ناهنجاریهای کروموزومی با ویژگیهای تومور بررسی میشود و با توجه به ارائه اطلاعات زیستشناسی مولکولی و کلینیکی قادر است ناهنجاریهای کروموزومی و بافتشناسی تومور را با توالیهای ژنومی مقایسه کند. لازم به ذکر است که اطلاعات این بانک داده به صورت دستی از مجلات توسط Fredrik Mitelman و همکارانش جمعآوری شده است.
۶۶-فصل سوم
بانک داده Recurrent Chromosome Aberrations Cancer از بانک Mitelman مشتق شده است و شامل تمامی تغییرات سیتوژنتیکی است که حد اقل در دو مورد مورفولوزی یکسان داشته باشند و در هر زمان تومورهای ثابت در مکان یکسان قابل اجرا میباشد.
Cancer Chromosome
به منظور فهرست کردن اطلاعات مربوط به ناهنجاریهای کروموزومی در سرطان که از تکنیکهای سیتوژنتیک مولکولی به دست آمدهاند و نیز برای ادغام کردن این اطلاعات با نقشههای ژنومی دو منبع تاسیس شده است.
۱: بانک داده SKY/M-FISH&CGH
۲: بانک داده Cancer Chromosome
هدف از تشکیل این بانک داده این است که به محقق این امکان را بدهد تا اطلاعات سیتوژنتیکی و کلینیکی حاصل از تحقیق را آنالیز کند و یا برای این بانک بفرستد. در بانک داده SKY/M-FISH&CGH امکان مقایسه اطلاعات سیتوژنتیکی و مولکولی فراهم شده است و میتوان بهطور همزمان کروموزومهای موجوداتی مثل موش و انسان را با توجه به رنگهای متفاوت مشخص شده در آنها بررسی کرده و ناهنجاریها را به سادگی نشان داد.
Protein Cluster
در این پایگاه داده کل پروتئینهای موجود در بانک refseq که از ژنوم کامل پروکاریوتها ،پلاسمیدها و فاژها بیان شدهاند بر اساس تشابه در توالی و عملکرد پروتئین گروهبندی میشوند.پروتئینها به صورت کامپیوتری در گروههایی بر مبنای امتیاز مربوط به همترازی آنها دستهبندی شده و هر دسته مربوط به یک شناسه منحصر به فرد میباشد.
Pop set
این پایگاه داده شامل توالیهای نوکلئوتیدی و پروتئینی است که به منظور بررسی خویشاوندی و سیر تکاملی یک جمعیت در این پایگاه گردآوری شده است.در این پایگاه با توجه به همترازی انجام شده بین توالیها کاربر قادر به توصیف وقایع سیر تکاملی و تنوع جمعیت توالیها میباشد. نتایج موجود در این پایگاه داده برای مطالعه و بررسی جهشها،روابط تکاملی در جمعیتها ،مورد استفاده قرار میگیرد.
Probe
در این پایگاه داده میتوان به روشهای مختلف در یک توالی ژن خاص،کاوشگر مورد نظر را جستجو کرد. انواع کاوشگرهای مورد جستجو در توالی شامل STS، RNAi ، RFLP و… میباشد.
dbGap
این پایگاه داده، نتایج مطالعات و بررسیهای انجام شده در مورد ارتباط بین ژنوتیپ و فنوتیپ را جمعآوری میکند. این مطالعات شامل مطالعات وسیع ژنومی، توالییابی، تستهای تشخیص مولکولی و نیز ارتباط بین ژنوتیپ و ویژگیهای غیر کلینیکی میباشد. این پایگاه داده شامل سه قسمت است. بخش اول Studyنام دارد و مقالاتی با موضوعات فوق در این بخش دیده میشود. بخش دوم Disease نام دارد و نام چند بیماری مهم ژنتیکی همراه با خصوصیت فنوتیپی آن در این قسمت آورده شده است. بخش سوم Advanced search نام دارد که جهت جستجوی پیشرفته و اختصاصی از این گزینه استفاده میگردد.
۶۷-پایگاههای اطلاعات اولیه توالیهای اسید نوکلئیک
BioSystems
The BioSystems database collects information on interacting sets of biomolecules involved in metabolic and signaling pathways, disease states, and other biological processes. BioSystems currently contains biological pathways from the Kyoto Encyclopedia of Genes and Genomes (KEGG) and the EcoCyc (Escherichia coli K-12 MG1655) subset of the BioCyc databases and is designed to accommodate other data in the future. BioSystems records link to related literature, genes, protein sequences, structures, chemical data, to related BioSystems. When available each record links to detailed diagrams and annotations for individual pathways on the Web sites of the source databases.
Bookshelf
The NCBI Bookshelf contains a collection of full-text books that can be searched online and that are linked to PubMed records through research paper citations within the text. The collection includes biomedical textbooks, other scientific titles, the NCBI News, and NCBI help manuals.
Conserved Domains
Conserved Domains is a database of protein domains represented by sequence alignments and profiles for protein domains conserved in molecular evolution. It also includes alignments of the domains to known three-dimensional protein structures in the MMDB database. The source databases for Conserved Domains are Pfam, Smart, and COG.
dbGaP
dbGaP (Database of Genotypes and Phenotypes) provides the results of studies that have investigated the interaction of genotype and phenotype including genome-wide association studies, medical sequencing, molecular diagnostic assays, as well as association between genotype and non-clinical traits.
dbVAR
dbVAR (Database of Genomic Structural Variation) contains information about large-scale genomic variation, including large insertions, deletions, translocations and inversions. dbVar also provides associations of defined variants with phenotype information.
Epigenomics
The Epigenomics database contains results of genome-wide studies on modifications of chromatin (histone modification, DNA methylation, DNAase footprinting) in various cell types that assay programmable changes that affect gene expression (epigenetics). Data from these studies may be displayed graphically on the genome sequence using the NCBI graphical sequence viewer.
EST
The EST database contains sequence records from the bulk EST (Expressed Sequence Tag) division of GenBank. These are typically short single-pass reads from cDNA libraries often generated as large survey project. Data from EST can be used to catalog expressed genes for a particular organ, tissue or cell type or general for a species, and compare expression levels of genes in various library sources.
Gene
Gene is a searchable database of genes, focusing on genomes that have been completely sequenced and that have an active research community to contribute gene-specific data. Information in Gene records includes nomenclature, chromosomal localization, gene products and their attributes (e.g., protein interactions), associated markers, phenotypes, interactions, and links to citations, sequences, variation details, maps, expression reports, homologs, protein domain content, and external databases.
Genome
The Genome database contains sequence and map data from the whole genomes of over 1000 species or strains. The genomes represent both completely sequenced genomes and those with sequencing in-progress. All three main domains of life (bacteria, archaea, and eukaryota) are represented, as well as many viruses, phages, viroids, plasmids, and organelles.
۶۸-فصل سوم
Genome Project
Genome Projects collects information on complete and in-progress large-scale sequencing, assembly, annotation, and mapping projects for cellular organisms. The database is organized as a set of organism-specific overviews that allow browsing and retrieving specific projects for that organism.
GEO Datasets
GEO Datasets stores curated gene expression and molecular abundance data sets assembled by NCBI from the Gene Expression Omnibus (GEO) repository of microarray data.
GEO Profiles
GEO Profiles is a database that stores individual gene expression and molecular abundance profiles assembled from the Gene Expression Omnibus (GEO) repository of microarray data.
GSS
The GSS database contains sequence records from the bulk GSS (Genome Survey Sequence) division of GenBank. These are the genomic equivalent of EST records; short single pass reads from gDNA libraries. Insert end and other reads from BAC and other large insert genomic libraries used to identify and assemble candidates for genome sequencing are common examples of GSS records.
HomoloGene
The HomoloGene database contains automatically generated sets of homologous genes and their corresponding mRNA, genomic, and protein sequence data from selected eukaryotic organisms. Potential homologs from other organisms are included through sequence similarity to UniGene clusters.
MeSH
MeSH (Medical Subject Headings) is the National Library of Medicine’s controlled vocabulary and classification system (ontology) used for indexing articles in PubMed. MeSH terminology provides a consistent way to retrieve information that may use different terminology for the same concepts. Searches in the Entrez MeSH database provide synonymous MeSH terms that can provide more useful results in PubMed. The MeSH database records show subheadings access the MeSH browser showing related concepts and hierarchical relationships among MeSH terms.
NCBI Web Site Search
NCBI Site Search is database of static NCBI web pages, documentation, and online tools. Searching this database is a quick way to find specialized online sequence analysis tools, back issues of newsletters, legacy resource description pages, sample code, and other miscellaneous resources.
NLM Catalog
The NLM Catalog contains records for books, journals, audiovisuals, computer software, electronic resources, and other materials in the National Library of Medicine (NLM) collections. The old Journals database was merged into the NLM Catalog database and the information once retrieved via Journals, is provided by the NLM Catalog. This includes data such as journal title, MEDLINE abbreviation, NLM ID, ISO abbreviation, or ISSN.
Nucleotide
Apart from sequence data in the EST (Expressed Sequence Tag) and GSS (Genome Survey Sequence divisions of GenBank, the Nucleotide database contains all the sequence data from GenBank, EMBL, and DDBJ, the members of the International Nucleotide Sequence Databases Collaboration (INSDC). Nucleotide also includes NCBI-curated Reference Sequences (RefSeqs), submitted assemblies and annotations from the Third Party Annotation (TPA) database, and nucleotide sequences extracted from structure records from the Protein Databank (PDB).
OMIA
OMIA (Online Mendelian Inheritance in Animals) is a database of genes, inherited disorders and traits in animal species (other than human and mouse). The database contains textual information and references, as well as links to relevant records from OMIM, PubMed, and Gene.
۶۹-پایگاههای اطلاعات اولیه توالیهای اسید نوکلئیک
OMIM
The OMIM (Online Mendelian Inheritance in Man) database contains review articles human genes, genetic disorders, and other inherited traits. OMIM articles provide links to associated literature references, sequence records, maps, and related databases.
PopSet
The PopSet database contains related nucleotide sequences that originate from comparative studies: phylogenetic, population, environmental (ecosystem), and mutational. Each record in the database is a set of nucleotide sequences representing the same molecule from the same species (population, mutation), different identifiable species (phylogenetic), or anonymous species from the same biological community (ecosystem).
Probe
Probe is a database of nucleic acid reagents designed for use in a wide variety of biomedical research applications including genotyping, gene expression studies, SNP discovery, genome mapping, and gene silencing. Probe records contain information on reagent distributors, probe effectiveness, and computed sequence similarities.
Protein
The Protein database contains amino acid sequences created from the translations of coding regions provided on nucleotide records in GenBank, EMBL, and DDBJ, the members of the International Nucleotide Sequence Databases Collaboration (INSDC) as well as those from coding regions on NCBI Reference Sequences and the Third Party Annotation (TPA) database records. Protein records are also imported from the outside protein-only data sources Protein Information Resource (PIR), SWISS-PROT, Protein Research Foundation (PRF). Protein sequences are also extracted from structure records from the Protein Data Bank (PDB).
Protein Clusters
Protein Clusters is a collection of related protein sequences (clusters) consisting of Reference Sequence proteins that are encoded by complete prokaryotic genomes as well those encoded eukaryotic organelle plasmids and genomes. The database provides easy access to annotation information, publications, domains, structures, external links, and analysis tools.
PubChem BioAssay
PubChem BioAssay is a database that contains bioactivity screens of chemical substances described in PubChem Substance. It provides searchable descriptions of each bioassay, including descriptions of the conditions and readouts specific to that screening procedure.
PubChem Compound
The PubChem Compound database contains unique, validated chemical structures (small molecules) that can be searched using names, synonyms or keywords. The compound records may link to more than one PubChem Substance record if different depositors supplied the same structure. Structures in PubChem Compounds are pre-clustered and cross-referenced by identity and similarity groups. Additionally, calculated properties and descriptors are available for searching and filtering of chemical structures. Compound records are linked to related PubChem Substance Records, PubMed citations, protein 3D structures, and biological screening results that are available in PubChem BioAssay.
PubChem Substance
The PubChem Substance database contains information on chemical substances including mixtures electronically submitted to PubChem by depositors. This includes any chemical structure information submitted, as well as chemical names, comments, and links to the depositor’s web site.
PubMed
PubMed is database of citations and abstracts for biomedical literature from MEDLINE and additional life science journals. Links are provided when full text versions of the articles are available through PubMed Central or other websites.
۷۰-فصل سوم
PubMed Central
PubMed Central (PMC) is the U.S. National Library of Medicine’s digital archive of life sciences journal literature. PMC contains full-text manuscripts deposited by authors or articles provided by the publisher.
SNP
The SNP (Single Nucleotide Polymorphism) database is a central repository for single nucleotide polymorphisms, microsatellites, and small-scale insertions and deletions. Both submitted SNPs and NCBI-produced non-redundant reference records (RefSNPs) that cluster reports of the same polymorphism from different sources are available. SNP also contains population-specific frequency and genotype data, experimental conditions, molecular context, and mapping information for both neutral polymorphisms and clinical mutations.
SRA
The SRA (Sequence Read Archive) contains sequencing data from the next generation sequencing platforms. SRA accepts and presents data from all current next-generation sequencing platforms including 454 (Roche), Illumina, SOLiD (Applied Biosystems), HeliScope, and Complete Genomics. Data can include sequence, quality scores, color values, and intensity graphs depending on the platform involved.
Structure
The Structure or Molecular Modeling Database (MMDB) contains experimental data from crystallographic and NMR structure determinations. The data for MMDB are obtained from the Protein Data Bank (PDB). Structure records link to bibliographic information, the sequence databases, and to the NCBI taxonomy. Cn3D, the NCBI 3D structure viewer, allows for easy interactive visualization of molecular structures from Entrez.
Taxonomy
The Taxonomy database contains the names and phylogenetic lineages of the more than 160,000 organisms that have molecular data in the NCBI databases. New taxa are added to the Taxonomy database as data are deposited for them. The taxonomy records include links to all molecular data for the organism or group as well as links to outside classification resources. The taxonomy provides the major controlled vocabulary for classifying molecular data across the Entrez system.
UniGene
UniGene is a database that provides automatically generated nonredundant sets (clusters) of transcript sequences, each cluster representing a distinct transcription locus (gene or expressed pseudogene). UniGene clusters also provide information on protein similarities, gene expression, cDNA clone reagents, and genomic location.
UniSTS
UniSTS is a comprehensive database of sequence tagged sites (STSs) derived from STS-based maps and other experiments. STSs are defined by PCR primer pairs and are associated with additional information, such as genomic position, genes, and sequences.