- 596
- ۱۴۰۲/۰۲/۰۶ - ۱۲:۲۴
- 815 بازدید
شرح فصل و نکات ویژه: * در این فصل مروری کلی بر جنبههای مختلف ژنومیکس ساختاری، مقایسهای و عملکردی خواهیم داشت. * مباحث مربوط به ژنومیکس مقایسهای و عملکردی در فصلهای بعد به تفصیل مورد بحث قرار میگیرند. * در این فصل انواع روشهای سکئونسینگ و پروژههای ژنوم معرفی میشوند. بسیاری از مباحث موجود در این فصل در فصول بعدی با تفصیل شرح داده شدهاند، به این دلیل برخی بخشها در این فصل به صورت خلاصه تدوین[…]
شرح فصل و نکات ویژه:
* در این فصل مروری کلی بر جنبههای مختلف ژنومیکس ساختاری، مقایسهای و عملکردی خواهیم داشت.
* مباحث مربوط به ژنومیکس مقایسهای و عملکردی در فصلهای بعد به تفصیل مورد بحث قرار میگیرند.
* در این فصل انواع روشهای سکئونسینگ و پروژههای ژنوم معرفی میشوند.
بسیاری از مباحث موجود در این فصل در فصول بعدی با تفصیل شرح داده شدهاند، به این دلیل برخی بخشها در این فصل به صورت خلاصه تدوین شدهاند. بحث اصلی این فصل پیرامون ژنومیکس ساختاری میباشد.
پیشنهاد مطالعاتی:
کتاب ”بیوانفورماتیک” نوشته ”محمدرضا نقوی” و ”محمدعلی ملبوبی” از انتشارات دانشگاه تهران کتابی جامع میباشد که به مبحث ژنومیکس بهصورت گستردهتر پرداخته است.
۲۶-فصل دوم
مدتهای طولانی است که پزشکان و دانشمندان میدانند ژنوم به عنوان مجموعه کامل اطلاعات ژنتیکی یک موجود زنده، یک منبع غنی از اطلاعات مربوط به عناوینی است که از متابولیسم پایه و مکانیسمهای نمو تا طول عمر و افزایش سن متفاوت هستند. هرچند، اندازه عظیم ژنوم انسانی ( جفت باز نوکلئوتیدی) نیاز به تغییر در روش دانشمندان برای تعیین توالیهای DNA را نشان میدهد و همچنین پیشرفتهای اخیر در بیوانفورماتیک به نوبه خود، نیاز به ابداع روش برای «استخراج» اطلاعات از توده توالی ژنومی حاصل از طرح ژنوم انسانی و گونههای مرتبط را مطرح میکنند. تکمیل موفقیتآمیز طرح ژنوم انسان اوج بیش از شش دهه تلاش ماندگار در بیولوژی مولکولی، ژنتیک و بیوشیمی است. شرح تاریخی زیر اشاره به یکایک وقایع مهمی دارد که منتهی به تعیین توالی کل ژنوم انسان شد.
۱۹۴۴- نقش DNA به عنوان ماده وراثتی نشان داده شد.
۱۹۵۳- مفهوم مارپیچ دوتایی مسلم شد.
۱۹۶۶- کد ژنتیکی مشخص شد.
۱۹۷۲- فناوری DNA نوترکیب ابداع شد.
۱۹۷۷- فناوری تعیین توالی DNA عملی شد.
۱۹۸۳- ژن بیماری هانتینگتون نقشهبرداری شد.
۱۹۸۵- واکنش زنجیری پلیمراز (PCR) ابداع شد.
۱۹۸۶- تعیین توالی DNA خودکار شد.
۱۹۸۶- ژن دیستروفی عضلانی دوشن شناسایی شد.
۱۹۹۰- طرح ژنوم انسان در ایالات متحده شروع شد.
۱۹۹۴- نقشهبرداری ژنتیکی انسان تکمیل شد.
۱۹۹۶- اولین نقشه ژنی انسان تهیه شد.
۱۹۹۹- چند شکلی تک نوکلئوتیدی مقدماتی شروع شد.
۱۹۹۹- اولین توالی یک کروموزوم انسانی (شمار ۲۲) تکمیل شد.
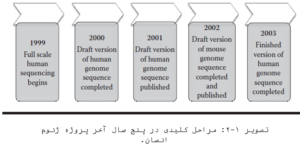
۲۰۰۰- «اولین پیشنویس» طرح ژنوم انسان تکمیل شد.
۲۰۰۳- تعیین توالی اولین ژنوم انسانی تکمیل شد.
همزمان با این پیشرفتها، تعیین توالی ژنوم صدها موجود زنده دیگر انجام شد، مثل Haemopilus influenzae (1995)، مخمر (۱۹۹۶)، Escherichia coli (1997)، Caenorhabditis elegans (1998)، Mycobacterium tuberculosis (1998)، برنج (۲۰۰۰)، Listeria monocytogenes (2001)، کوروناویروس SARS (2003)، موش صحرایی (۲۰۰۴) و شامپانزه (۲۰۰۵).
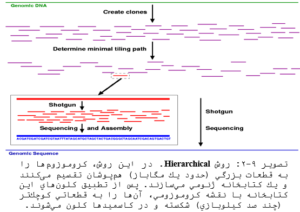
دو گروه مسئول تعیین توالی ژنوم انسان بودند. گروه اول “اجلاس تعیین توالی ژنوم انسان” بود که از تعیین توالی “Shotgun sequencing سلسله مراتبی” استفاده نمود (تصویر ۹-۲) و کار خود را از سال ۱۹۹۰ شروع کرد. کل ژنوم به قطعاتی در حدود kb 200-100 تجزیه شد و این قطعات در داخل کروموزومهای ساختگی باکتریایی (BACs) قرار داده شدند. سپس این BACها از طریق جستوجوی توالیهای نشانگری به نام جایگاههای توالی که موقعیت آنها قبلاً تعیین شده بود، در BACهای مجزایی قرار داده شدند. در مرحله بعد، کلونهای BACs به قطعات کوچک شکسته شدند. سپس هر قطعه تعیین توالی شد و از الگوریتمهای کامپیوتری برای سازماندهی توالیهای سازگار اطلاعات حاصل از قطعات همپوشان برای جمعآوری کامل
۲۷-ژنومیکس
این توالیها استفاده گردید. گروه دوم تیم Celera بود که کار خود را از سال ۱۹۹۸ شروع کرده و از روش “shotgun sequencing کل ژنوم” استفاده نمود (تصویر ۷-۲). در این روش به جای اینکه ابتدا کلونهای موجود در کتابخانه ژنوم را به صورت شمارشی مرتب کرده و سپس تعیین توالی کنند، ابتدا BACها را تعیین توالی کرده و سپس از یک الگوریتم کامپیوتری برای تعیین ترتیب قطعههای کلون شده استفاده میشود. در سال ۲۰۰۳ “اجلاس تعیین توالی ژنوم” اعلام کرد ۹۰% یوکروماتین ژنوم انسان به پایان رسیده است و در همین تاریخ شرکت Celera اعلام کرد ۹۳% یوکروماتین ژنوم انسان را تعیین توالی کرده است.
ژنومیکس علم مطالعه ژنوم است، بیراه نگفتهایم اگر ادعا کنیم شاهراه اصلی بیوانفورماتیک ژنومیکس میباشد و میتوان بسیاری از مباحث بیوانفورماتیک را حول بحث ژنومیکس بیان کرد. در این فصل به مباحث مختلف حوزه ژنومیکس اشاره خواهیم کرد، این بحث را میتوانیم ” Pre-genomic to Post-genomic Era” بنامیم. ژنومیکس به سه بخش کلی با عناوین ژنومیکس ساختاری، ژنومیکس مقایسهای و ژنومیکس عملکردی تقسیم شده است که در این فصل مبحث ژنومیکس ساختاری به تفصیل شرح داده شده است اما مباحث ژنومیکس مقایسهای و عملکردی به علت همپوشانی مباحث در فصلهای بعدی به صورت خلاصه شرح داده شدهاند. درمطالعه ژنومیکس تعداد بسیار زیادی ژن و یا تمام ژنهای جاندار بهطور هم زمان مورد مطالعه قرار میگیرد. نقشه یابی ژنوم و همچنین توالییابی ژنوم از چالشهای اصلی و ابتدایی این حوزه از علم میباشد و سپس تجزیه و تحلیل دادهها چالشهای بعدی میباشد.
۲۸-فصل دوم
۱-۲ ژنومیکس ساختاری
در ژنومیکس ساختاری تجزیه و تحلیلهای اولیه ژنوم، شامل: ترسیم نقشههای فیزیکی و ژنتیکی، شناسایی ژنها، مستندسازی ژنها و مقایسه ساختارهای ژنوم صورت میگیرد. مباحثی که در رابطه با ژنومیکس ساختاری در ادامه مطالعه خواهید کرد به این شرح میباشد: نقشهیابی ژنوم، توالییابی ژنوم، تفسیر ژنوم، انتولوژی ژن، مستندسازی خودکار ژنوم، مستندسازی پروتئینهای فرضی، ساختار ژنوم، گروههای پروتئین ارتولوگ، نواحی رمز کننده، نواحی غیررمز کننده، تعداد ژنها در ژنوم و اقتصاد ژنوم. نیم قرن پس از ارائه مدل ساختمانی DNA توسط واتسون و کریک (۱۹۵۳ م)، پروژه ژنوم انسان به عنوان بزرگترین پروژه پژوهشی بشر با صرف بیش از ۲ میلیارد دلار و ۱۲ سال (در سال ۲۰۰۳ م) به اتمام رسید.
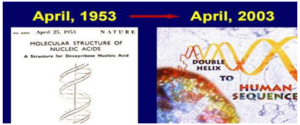
تصویر ۲-۲: اعلام اختتام پروژه ژنوم انسان ۵۰ سال پس از ارائه مدل واتسون و کریک.
در حال حاضر، بدون احتساب ویروسها، بیش از ۴۵۰۰ پروژه ژنوم گونههای مختلف پروکاریوتی و یوکاروتی در حال انجام است. تا کنون بیش از ۳۰۰ پروژه ژنوم (بهجز ویروسها) اتمام یافته است و تقریبا هر ماه پایان دو پروژه اعلام میشود. با در اختیار داشتن فناوریهای جدیدتر (ادامه این بخش را ببینید)، سرعت انجام پروژههای ژنوم به مراتب بیشتر خواهد شد. اندازه کوچکترین ژنوم یک موجود تک سلولی مثل مایکوپلاسما ژنیتالیوم ۶۰۰ هزار جفت باز و ژنوم موجوداتی مثل موش و انسان حدود سه میلیارد جفت باز است. این ژنومها به صورتهای مختلف حلقوی و خطی میباشند که به حالت بستهبندی شده در داخل سلول در ساختارهایی به نام کروموزمها قرار دارند. بنا به اندازهی ژنومها، شکل ژنوم و چرخه زندگی موجود، راهبردهای متفاوتی برای تهیه توالی ژنومها به کار گرفته میشود.
از نظر ماهیت، اطلاعات حاصل از پروژههای ژنوم در سه گروه قابل تقسیم هستند:
۱- نقشههای و توالیهای مربوط به ژنومها که به ژنومیکس (Genomics) مشهور است.
۲۹-ژنومیکس
۲- ژنهای قابل رونویسی و توالی آنها که به ترانس کریپتومیکس (Transcriptomics) مشهور است.
۳- پروتئینهای ابراز شده و توالی آنها به پروتئومیکس (Proteomics) مشهور است.
در این بخش سعی شده است تصویری عمومی از پروژههای ژنوم و نحوه دسترسی به اطلاعات گروه اول آورده شود. گروه دوم و سوم در بخشهای بعدی به تفصیل تشریح شدهاند.
در ادامه، مباحث مربوط به ژنومیکس ساختاری را به ترتیب زیر دنبال کنید:
۱- نقشهیابی ژنوم ۷- ساختار ژنوم
۲- توالییابی ژنوم ۸- گروههای پروتئینی ارتولوگ
۳- تفسیر ژنوم ۹- نواحی رمز کننده
۴- انتولوژی ژن ۱۰- نواحی غیررمز کننده
۵- مستندسازی خودکار ۱۱- تعداد ژنها در ژنوم
۶- مستندسازی پروتئینهای فرضی ۱۲- اقتصاد ژنوم
۱-۱-۲ نقشهیابی ژنوم
اولین گام در فهم ساختار ژنوم نقشهیابی آن است. مکانهای نسبی ژنها، جهشها یا صفات مورفولوژیکی بر روی کروموزومها شناسایی میشوندو لازم به ذکر است معمولا کیفیت نقشهیابیها پایین است. از گذشتهها نقشههای ژنومی برای تعیین محل لوکوسهای تعیین کننده صفاتی خاص یا نشانگرها (Markers) به کار میرفتهاند. با آغاز پروژههای ژنوم، توجه پژوهشگران به استفاده از این نقشهها برای علامتگذاری نقاط معین (Landmarks) در ژنومها جلب شد تا بتوانند از آنها برای تشخیص نقاط مورد مطالعه در سطح توالی استفاده کنند. نشانگرها هر گونه صفتی اعم از صفات ظاهری (نشانگرهای مورفولوژیک) تا قطعات ژنومی یا ژنها (نشانگرهای مولکولی) را شامل میشوند. صفاتی مانند تعداد پرچم، رنگ بذر، مقاومت به بیماری از انواع نشانگرهای مورفولوژیک هستند. نشانگرهای مولکولی بنا به ماهیت آنها نامگذاری شدهاند. RFLP، RAPD، AFLP، VNTR، SSCP و SNP انواعی از این نشانگرها هستند که در دهههای
۳۰-فصل دوم
اخیر رواج فراوانی یافتهاند (برای اطلاعات بیشتر در رابطه با تنوع زیستی فصل ۱۳ را ببینید).
الف) نقشههای لینکاژی (ژنتیکی)
از سال ۱۹۱۰، ژنتیکدانان دریافته بودند که بین برخی صفات پیوستگی وجود دارد. با این استدلال که میان فاصله بین نقاط و احتمال کراسینگاور (Crossing over) ارتباط مستقیمی هست، اقدام به تخمین فاصله بین لوکوسهای مربوط به صفات پیوسته به هم نمودند. این شیوه مبنای تهیه نقشههای ژنومی شد که از طریق تجزیه و تحلیل اطلاعات ژنتیکی به دست میآمد و لذا نقشههای ژنتیکی نامیده شدند. این نقشهها، ترتیب قرار گرفتن لوکوسها و فاصله تخمینی بین آنها را نشان میدهند. واحد فاصله در این نقشهها، سانتیمورگان است. امروزه میدانیم به دلیل وجود نقاط حساس و مقاوم به شکنندگی یا کراسینگآور نقشههای ژنتیکی دقت لازم را ندارند.
– موقعیتهای نسبی نشانگرهای ژنتیکی براساس چگونگی به ارث رسیدن آنها شناسایی میشوند.
– یک واحد سانتیمورگان یعنی یک درصد از کل حوادث نوترکیبی به هنگام جدا شدن دو نشانگر ژنتیکی.
ب) نقشههای فیزیکی
در این گونه نقشهها با استفاده از مشاهده و یا محاسبه، فاصله بین دو نقطه کروموزومی بهطور فیزیکی نشان داده میشود. این نقشهها طی مطالعات سیتوژنتیکی یا کروموزومی از مشاهده شکستگیهای طبیعی کروموزومها ویا تحت تاثیر اشعهها به دست میآیند. بنا به روش مورد استفاده میزان تمایز (Resolution) بین دو نقطه تفاوت میکند. مثلا نقشههای سیتوژنتیکی که از مشاهده الگو و یا کم و زیاد شدن باندهای رنگآمیزی شده کروموزومها به دست میآیند، قدرت تمایز کمتری دارند. نقشههای (Radiation Hybrid maps) RH براساس میزان اشعه تابانده شده یا سانتیگری (CR) تعیین میشوند. هر چقدر مقدار تشعشع به سلولهای در حال مطالعه بیشتر باشد، شکستگیهای بیشتر و نزدیکتر به هم ایجاد میشود و در نتیجه قدرت تمایز نقشه بالا میرود. در این روش سلولهای اشعه دیده با سلولهای موجودی دیگر دو رگ میشوند. سپس در سلولهای حاصل به مطالعه نوع شکستگی و تاثیر آن میپردازند. قدرت تمایز این نقشهها تا ۵۰ کیلوباز میرسد.
۳۱-ژنومیکس
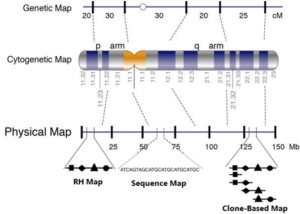
تصویر ۴-۲: نمایی از نقشههای ژنتیکی و فیزیکی مورد نیاز پروژههای ژنوم.
در سالهای اخیر نقشههایی با همین قدرت تمایز با استفاده از تعیین توالی دو انتهای کلونهای BAC به دست میآید که این نقشهها را STC map مینامند. در مقایسه، نقشههای توالی که بر مبنای تعیین توالی تهیه میشوند، بالاترین تمایز و دقت را دارند؛ زیرا در این نقشهها میتوان فاصله بین دو نقطه را برحسب جفت باز (bp) تعیین کرد. به هر حال، مقدمه تعیین توالی ژنوم، تهیه نقشههای ژنتیکی و فیزیکی است.
– در نقشههای فیزیکی موقعیت مکانهای قابل تفکیک و مشخص در ژنوم بدون توجه به الگوی توارث آنها تعیین میشود.
– نقشههای فیزیکی با استفاده از روشهای پیمایش کروموزومی که مبتنی بر کاوشگرهای برچسبدار شده برای دورگه گیری با قطعات کلون شده DNA است به دست میآیند.
ج) نقشههای سیتوژنتیکی
هر کروموزوم بعد از رنگآمیزی الگوی ویژه باندی (تیره و روشن) را نشان میدهند. در این نوع نقشهبرداری فواصل بین دو کروموزوم نسبی است و کیفیت نقشهها پایین میباشد.
تهیه کتابخانه ژنومی
در حالی که تلاشهای در حال انجام برای تهیه نقشههای ژنومی ادامه یافته و شتاب بیشتری میگرفت، به منظور فراهم آوردن امکانات برای تعیین توالی و رفع مشکلات آن، روشهای تهیه کتابخانههای ژنومی نیز توسعه یافت. بدین لحاظ، آزمایشگاههای شرکت کننده در پروژههای ژنوم نسبت به تهیه کتابخانههای ژنومی اقدام نموده و آنها را با یکدیگر رد و بدل مینمودهاند. انواع مشهور کتابخانههای ژنومی در جدول زیر آورده شدهاند.
۳۲-فصل دوم
جدول ۱-۲: اطلاعات مقایسهای کتابخانههای ژنومی به کار رفته در پروژههای ژنوم.
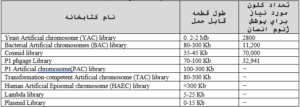
لازم به ذکر است برای تهیه کتابخانههای موجودات مختلف و به ویژه انسان سعی برآن است که فرد خاصی را در نظر نگیرند. به این ترتیب توالی عام (Blueprint) مربوط به یک گونه خاص (و نه فرد) تهیه میشود که میتواند پایه مطالعات ژنومی واقع شود.
۲-۱-۲ توالییابی ژنوم
سه رویکرد اصلی برای توالییابی کل ژنوم وجود دارد.
– روش شاتگان (Shotgun Approach)
– روش سلسلهمراتبی (Hierarchical Approach)
– تعیین توالی در مقیاس انبوه (Large-Scale Sequencing)
از آنجا که محدودیتهای تکنیکی موجود امکان تعیین توالی یک ژنوم یا یک کروموزوم بهطور پیوسته را امکان پذیر نمیکند، تلاش های زیادی برای افزایش اندازه قابل توالی یابی انجام گرفته است. در روشهای معمول، میزان توالی تعیین شده در هر واکنش حدود ۳۵۰ تا ۷۰۰ جفت باز است. به ندرت میتوان با روشهای اتوماتیک تا ۱۰۰۰ جفت باز را تعیین توالی نمود. به دلیل چنین محدودیتی نیازمند به تهیه کتابخانههای ژنومی که در آن ناقلها توالیهای کوتاه (به طول ۱ تا ۵۰ کیلوباز) را حمل میکنند هستیم (جدول ۱-۲). کوچک شدن اندازه قابل توالییابی، بهره گیری از راهبردهایی برای کنار هم چیدن توالیهای به دست آمده در جهت به دست آوردن قطعات بزرگتر یا تکه توالی (conting) را ضروری میسازد. براساس این راهبردها، سه راهبرد برای تعیین توالی ابداع شده است: راهبرد اول، روشی کلاسیک بوده و از ابتدا در پروژههای ژنوم به کار میرفته است. در این روش با کوچک و کوچک کردن قطعات، اقدام به تعیین توالی شده و سپس با استفاده از توالی های هم پوشان و نقشه ها قطعات سرهم می شوند تا توالی طول کامل هر کروموزوم بدست آید. این راهبرد بسیار پرهزینه بوده و اغلب با بنبستهای تکنیکی مواجه بوده است.
۳۳-ژنومیکس

تصویر ۵-۲: الکتروفورز DNAهای نشاندار شده با موارد رادیو اکتیو به منظور سکوئنسینگ.
راهبرد دوم، مبتنی بر روش شلیک تصادف (Shot gun) در دو دهه گذشته ابداع شد و هماکنون بسیار رواج یافته است. در راهبرد اول و دوم، اساس روش تعیین توالی سانگر است.

راهبرد سوم، برای تعیین توالی در مقیاس انبوه معرفی شده است که روش کار آن در ادامه فصل میآید. دانستن روشها به شما در فهم و دنبال نمودن اطلاعات کمک زیادی میکند. نمایشی از نتایج تعیین توالی به روش دستی با استفاده از رادیوایزوتوپها (تصویر ۵-۲) و اتوماتیک با استفاده از مواد فلورسانت (تصویر ۶-۲) که در هر دو مورد از روش سانگر استفاده میشود در فوق آمده است.
الف) توالی یابی شاتگان (Shotgun Approach) (BAC-based sequencing)
در این روش، DNA کروموزمها در کتابخانههای BAC قطعه قطعه شده و جای داده میشود. قطعات موجود در BAC اندازههایی بین ۴۵ تا ۳۰۰ کیلوباز دارند که حدی متوسط بین کتابخانههای کاسمیدی و YAC است. بعلاوه، در این روش، نیاز چندانی به نقشههای ژنتیکی و فیزیکی نیست، بلکه میتوان با تعیین توالی دو انتهای هر قطعه کلون شده در ناقل BAC و تعیین موقعیت آن در روی کروموزوم یک نقشه STC به دست آورد که خود مبنای توالی دارد. در قدم بعدی، هر کلون BAC به قطعات کوچکتر همپوشان تقسیم کرده و در ناقلها پلاسمیدی جای میدهند. سپس بهطور تصادفی کلونهایی را انتخاب و تعیین توالی میکنند. این کار آنقدر ادامه مییابد تا توالیهای هم پوشان متعددی برای یک منطقه به دست آید (شکل ۷-۲). بدین لحاظ، این روش را روش شلیک تصادفی (Shot-gun seq) نیز مینامند. برای هر سری تعیین توالی به این روش، بهطور معمول یک منطقه ۱۰تا ۳۰ بار تعیین توالی میشود. ولی از آنجا که زمان و هزینهای برای انتخاب کلونها صرف نمیشود و روش تا حد زیادی قابلیت خودکار شدن دارد، در مجموع این روش سریعتر و ارزانتر تمام میشود.
۳۴-فصل دوم
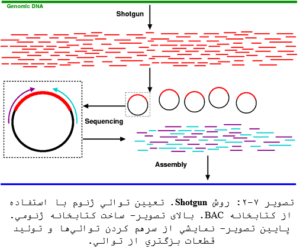
هدف: سر هم کردن توالیهای کوچک به دست آمده از روش پایاندهی زنجیره و ایجاد توالی اصلی است.
– به توالیهای کوچک کانتیک میگویند و با اتصال کانتیکها قطعات اسکفولد ایجاد میشوند.
– توسط مناطق همپوشان بین کانتیکها توالی اصلی را مییابیم.
مراحل روش شات گان:
۱- استخراج ¬DNA 2- شکستن DNA با امواج صوتی به قطعات کوچک ¬ ۳- شناسایی قطعات مناسب برای توالییابی (قطعات الکتروفورز میشوند آنهایی که بین ۶/۱ تا ۲ کیلو باز هستند از ژل جدا میشوند) ¬ ۴- کلون کردن قطعات به منظور تکثیر (برای اطمینان از حضور قطعه در کلون PCR انجام میدهند) ¬ ۵- جداسازی و خالصسازی قطعه کلون شده ¬ ۶- توالییابی با روش اتومات ختم زنجیره ¬ ۷- سر هم کردن کانتیکهای ۶/۱ تا ۲ توسط قسمتهای همپوشان (اگر کل طول توالیهای همپوشان ۶ تا ۱۰ برابر باشد میتوان اطمینان پیدا کرد). از نرمافزارهای Phred و Phrap برای مقایسه قطعات توالییابی شده، پیدا کردن مناطق همپوشان و مرتب کردن آنها میتوان استفاده کرد. اگر قسمتی توالییابی نشده بود
۳۵-ژنومیکس
با استفاده از بخشی که توالی آن را شناختیم آن قسمت را توالییابی میکنیم (به این کار Fishing میگویند).
ب) توالی یابی سلسلهمراتبی (Hierarchical Approach) (Map-based sequencing)
این روش، تعیین توالی از بالا به پایین (Top-down seq) و تعیین توالی کلون به کلون (Clone-by-clone seq) نیز نامیده میشود. در این روش، کروموزومها را به قطعات نسبتا بزرگی (حدود یک مگاباز) همپوشان تقسیم کرده و یک کتابخانه ژنومی از نوع YAC (Yeast Artificial Chromosomes) میسازند. پس از تطبیق کلونهای این کتابخانه با نقشه کروموزومی، آنها را به قطعاتی کوچکتر (چند صد کیلوبازی) شکسته و در کاسمیدها کلون مینمایند. سپس کلونهای کاسمیدی را در کتابخانههای لامبدا (۱۰ تا ۲۵ کیلوبازی) و یا پلاسمیدی (۱ تا ۱۵ کیلوبازی) تقسیم میکنند. کلونهای اخیر قابل توالییابی هستند.
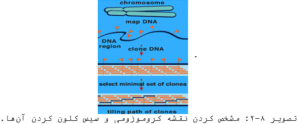
پس از تعیین نقشه یک کروموزوم و تعیین توالی این کلونها، با کنار هر چیدن توالیها، مسیر معکوس از کلون پلاسمیدی به کاسمیدی، به YAC و بالاخره به کروموزم دنبال میشود. به عبارت دیگر، تکه توالیها بزرگ و بزرگتر شده و به اندازه کروموزوم برسند. این روشی بود که تا سال ۱۹۹۸ بهطور عمده در تعیین توالیهای ژنومها به کار میرفت. اما در حین انجام پروژه ژنوم انسان (اولین پروژه ژنوم) اشکالات زیر به خوبی مشخص گردید.
* غیرقابل همسانهسازی برخی توالیها و در نتیجه باقی ماندن فاصلهها در برخی مناطق کروموزومی.
* غیر ممکن بودن تعیین توالی برخی مناطق ژنومی با ساختارهای خاص بویژه در توالیهای غنی از G/C.
* میزان بالای بازترتیبی (rearrangement) همسانههای کتابخانههای ژنومی YAC.
دستیابی به فناوری ساخت کتابخانههای BAC تا حد زیادی این سه مشکل را حل نمود. زیرا تا به حال بازترتیبی در همسانههای BAC نشان داده نشده است. علاوه بر آن، تعیین توالی دو انتهای هر همسانه با استفاده از آغازگرهای (primers) مبتنی بر توالی ناقل اجازه میداد تا همسانههای ارتباط دهنده فواصل خالی با استفاده از توالیهای هم پوشان (شکل زیر) به راحتی پیدا شوند. بدین لحاظ توالی دو انتهای این همسانهها را توالیهای نشانمندساز پیوند دهنده (Sequence Tag Connectors) یا STC نامیدند. همانطور که در بالا گفته شد، تهیه نقشه بر مبنای توالیهای دو انتهای همسانههای کتابخانههای BAC را STC map نامیدند که علاوه بر پر کردن فواصل خالی، نقشهای با قدرت تمایز مطلوب (تا ۵۰ کیلو باز) را فراهم میآورد.
۳۶-فصل دوم
– این نوع توالییابی نسبت به روش شات گان کندتر و پرهزینهتر میباشد اما بعد از تهیه نقشه تجمع توالیها آسانتر است.
– توالییابی سلسله مراتبی شبیه شاتگان است اما در مقیاس کوچکتری از ژنوم انجام میشود، یعنی ژنوم به صورت بخش به بخش توالییابی میشود و سپس نتایج به هم متصل میشوند تا به کل توالی دست یافت.
۱- کروموزومها با استفاده از راهبرد نقشهیابی فیزیکی نقشهیابی میشوند.
۲- سپس قطعات ۱۰۰ تا ۳۰ کیلو بازی به دست آمده در حاملهای BAC کلون میشوند.
۳- براساس نقشهیابی فیزیکی مکانها ترتیب BACها تعیین میشود. BACهای مرتب شده توالییابی میشوند.
۴- هر کلون BAC با روش شاتگان توالییابی میشود.
توالی یابی شاتگان برای توالی یابی ژنومهای کوچک مفید و کاراست ولی برای ژنومهای پیچیده یوکاریوتی خطاپذیر است و فاصلههای فراوانی در توالیهای حاصله به هنگام سرهم کردن کردن قطعات هم پوشان باقی میگذارد. امروزه برای توالییابی ژنومهای بزرگ (یوکاریوتها) از ترکیب دو روش استفاده میشود اگر پژوهشگران مجبور به انتخاب یکی از دو روش برای بررسی ژنوم یوکاریوتی باشند روش سلسله مراتبی کاراتر میباشد.
مشکلات روش سلسله مراتبی
– مشکل آلودگیها توسط توالیهای ناقل که توسط برنامهای قبل از سر هم کردن توالیها میتوان آلودگیها را از بین برد.
– مشکل قسمتهای تکراری توسط برنامههایی مثل Repeat Masker و روش Forward-Reverse Constraint میتوان از بین برد. Vec Screen برنامهای اینترنتی برای شناسایی توالیهای حامل باکتریایی در بین توالیهای تعیین توالی شده.
۳۷-ژنومیکس
– مشکل قسمتهای تکراری توسط برنامههایی مثل Repeat Masker و روش Forward-Reverse Constraint میتوان از بین برد. Vec Screen برنامهای اینترنتی برای شناسایی توالیهای حامل باکتریایی در بین توالیهای تعیین توالی شده. TIGR Assembler در پایگاه TIGR وجود دارد و برای سرهمبندی توالیهای بزرگ از محدودیت Forward-Reverse استفاده میکند. در این نرم افزار سرهمبندی توسط الگوریتم اسمیت– واترمن انجام میشود. ARACHANE برنامهای برای سرهمبندی توالیهای کل ژنوم میباشد که از یک روش تجربی استفاده میکند.
ج) تعیین توالی در مقیاس انبوه (Large-Scale Sequencing)
تکنولوژی توالییابی الکتروفورز موئینه (CE) سنگر بهطور گسترده در آزمایشگاههای سراسر دنیا بهکار گرفته شده است، اما محدودیتهای داخلی در توان عملیاتی، مقیاسپذیری، سرعت و تفکیکپذیری مانعی است که موجب جلوگیری دانشمندان از دسترسی به اطلاعات اساسی مورد نیاز شده است. برای غلبه براین موانع، یک تکنولوژی جدید (next-generation sequencing (NGS)) مورد نیاز است، این تکنولوژی اساساً یک رویکرد متفاوت در توالییابی است که کشفهای متعددی را باعث شده و تحولی را در علوم ژنومیک ایجاد کرده است. شرکت Roche اخیرا فناوری جدیدی را معرفی کرده است که مدعی است با استفاده از دستگاه Genome Sequencer FLX و گرایش Sequencing 454 (مبتنی بر روش Pyrosequencing) میتوان با یک پژوهشگر و در عرض ۳ روز با صرف حدود ۱۰۰ هزار دلار تا ۵۰ مگاباز (محدوده اندازه ژنومهای باکتریایی) را تعیین توالی نمود.
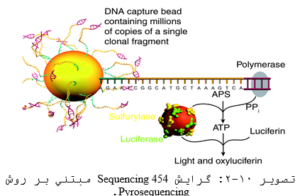
در این روش ابتدا یک کتابخانه ژنومی متصل به ذرات (و نه ناقل) ایجاد میشود که در حفرات بسیار ریز (در حد پیکومتر) توزیع میشوند. سپس واکنش pyrosequencing در حضور هر یک از بازها بهطور مجزا انجام میشود. ثبت تصاویر از نقاط فلورسانس در هر دور و تجزیه و تحلیل توالیهای کوتاه (۲۰۰ تا ۳۰۰ جفت باز) توسط نرمافزار باعث تولید توالی سرهمبندی شده با طولهای بلند (۵۰ مگاباز) و با درجه صحت بالا میشود. گذشت پنج سال از ارائه تکنولوژی NGS (بهعنوان تحول اساسی در مسیر استخراج اطلاعات ژنتیکی از سیستمهای بیولوژیکی) جنبههای بیحد و حصری از ژنوم، رونویسی و اپیژنوم گونهها را آشکار کرده است. این قابلیت تعدادی از موانع مهم را برطرف کرده و حوزههایی از علوم و تحقیقات در مورد بیماریهای انسان تا کشاورزی و علوم تکاملی را توسعه داده است. مفهوم تکنولوژی NGS اساساً شبیه به CE میباشد، بازهای قطعات کوچک DNA بهوسیله سیگنالهای منتشر شده در زمان سنتز هر قطعه (از روی رشته DNA الگو) تشخیص داده میشود. NGS این فرآیند را بهجای یک یا تعداد کمی از قطعات DNA، با میلیونها واکنش موازی انجام میدهد. این پیشرفت، تعیین توالی سریع مناطق بزرگی از جفت بازهای DNA در سراسر ژنوم را مقدور ساخته است.
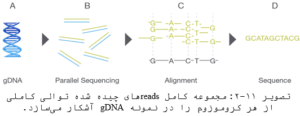
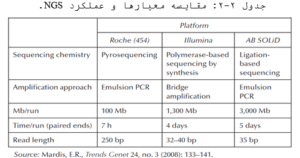
gDNA در ابتدا به کتابخانهای از قطعات کوچک تقسیم میشود که بهدرستی و بهصورت یکسان به میلیونها واکنش موازی توالییابی میشود. سپس رشتهای که توالییابی میشود (reads) با استفاده از یک ژنوم مرجع شناخته شده (توالییابی مجدد) و یا در غیاب ژنوم مرجع (توالییابی جدید) دوباره چیده میشود. مجموعه کامل readsهای چیده شده توالی کاملی از هر کروموزوم را در نمونه gDNA آشکار میسازد. پلتفرم های مختلفی از NGS توسط شرکتها ارائه شده است که در برخی از معیارها همچون طول reads و یا مدت زمان انجام کار با هم تفاوت دارند که برخی از این موارد در جدول زیر آمده است.
یک محقق برای انجام مطالعات دقیقتر میتواند پوشش ایجاد شده برای نوع خاصی از آزمایش را تغییر دهد. اصطلاح پوشش بهطور عمومی به میانگین تعداد خواندنهای توالی اشاره میکند که برای هر باز در نمونه DNA در یک ردیف قرار میگیرند. برای مثال، کل ژنومی که با پوشش ۳۰x توالییابی شده است به این معنی است که بهطور میانگین، هر باز در ژنوم ۳۰ بار خوانده شده است.
طبیعت دیجیتال NGS یک دامنه پویای نامحدود را حمایت میکند و حساسیت بسیار بالایی را برای استفادههای کمی از قبیل آنالیز بیان ژن فراهم میآورد. دانشمندان با NGS میتوانند فعالیت RNA را با دقتی بالاتر از روشهای مبتنی بر microarray کمی سازی کنند که برای درک تغییرات ظریف بیان ژن که در ارتباط با فرآیندهای بیولوژیکی بسیار اهمیت دارد.
توالییابی کل ژنوم
۳۹-ژنومیکس
حتی برای ژنوم ویروسی نسبتاً جمع و جور با ژنهای بههم فشرده، تعیین توالی کل ژنوم با استفاده از تکنولوژی مبتنی بر CE سنگر نیازمند زمان و منابع زیادی است. برای مثال، توالییابی De Novo کل ژنوم ویروس آبله گاوی (DNA پیچیده و بزرگ ویروسی در حدود ۲۰۰ کیلو باز) با استفاده از روش مبتنی بر CE، حدود ۴۰۰۰ واکنش تعیین توالی را به همراه دارد (با پوشش x10 و طول خواندن bp500) که هر کدام در لولهها یا چاهکهای جداگانه انجام میشود. اما پروژه تعیین توالی مشابه با استفاده از تکنولوژی NGS میتواند تنها در عرض چند روز و با یک دوره توالییابی و با پوشش x30 یا بیشتر انجام گیرد.
چالش ما برای تعیین توالی ژنومهای کوچک بهخاطر در دسترس نبودن مرجع ژنومی برای بسیاری از گونههاست. این بدان معنی است که تعیین توالی کل ژنوم بایستی بهصورت De Novo انجام گیرد. کیفیت پوشش مجموعه دادههای تعیین توالی
De Novo وابسته به کیفیت contigها (توالیهای پشت سرهم ایجاد شده توسط خواندن توالی همپوشانی شده) میباشد. سایز و تداوم کانتینگ (contig) بر روی تعداد شکافهای موجود در داده اثر میگذارد. مشکل تعیین توالی De Novo این است که طول خواندن کوتاه که با NGS ایجاد شده است میتواند منجر به تعداد بالایی از شکافها (مناطقی که چیدمان خواندن ندارند) شود. این موضوع بهخصوص در مورد مناطقی از ژنوم که حاوی عناصر تکراری میباشند نیز صدق میکند. برای غلبه بر این چالش بعضی از پلت فرمهای NGS، پروتکل تعیین توالی (Paired-End) PE را پیشنهاد کردهاند (تصویر ۱۲-۲)، در این پروتکل هر دو انتهای قطعه DNA تعیین توالی میشود و عکس زمانی است که فقط از یک انتها تعیین توالی صورت میگیرد. خواندن PE منجر به یک چیدمان بهتر در اطراف نواحی با توالی تکراری شده و با پر کردن شکافها در توالی مورد توافق کانتینگهای بلندتری را برای تعیین توالی De Novo ایجاد میکند که منجر به پوشش سراسری کاملی میشود.
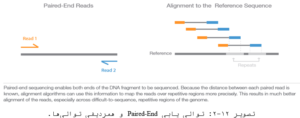
توالییابی هدفدار
با تعیین توالی هدفدار، تنها زیر گروهی از ژنها یا مناطق مشخص شده در ژنوم توالییابی میشوند و اینکار به دانشمندان اجازه میدهد تا بر روی زمان، هزینه و دادههای مناطقی از ژنوم که بیشتر مدنظر هستند،
۴۰-فصل دوم
متمرکز شوند. به منظور تعیین توالی هدفدار دو روش متفاوت (توالییابی آمپلیکون و غنیسازی هدف) برای ساخت کتابخانه وجود دارد.
بانکهای آنلاین دادههای NGS و آنالیز دادهها:
پایگاه SRA که در سایت NCBI قرار دارد محلی میباشد که دادههای حاصل از NGS در آن ذخیره میشوند و پژوهشگران به این دادهها دسترسی کامل دارند. همچنین پایگاه ENA که در سایت EBI به آدرس ebi.ac.uk/ena قرار دارد دادههای مختلف سکوئنسینگ از جمله دادههای NGS را در خود دارد. پایگاه TRACE به آدرس trace.ddbj.nig.ac.jp که در ژاپن واقع میباشد نیز حاوی دادههای NGS میباشد. یک پایگاه به نام deepBase وجود دارد که دادههای NGS مربوط به microRNAها در آن ذخیره میشود. پایگاه GEO به جمعآوری دادههای بیان ژن میپردازد و اکثر دادههای موجود در این پایگاه مربوط به دادههای حاصل از میکرواری میباشند اما دادههای مربوط به RNAseq نیز در این پایگاه موجود و قابل دریافتند. پایگاهی تحت عنوان TCGA وجو دارد که به جمع آوری دادههای NGS مربوط به سرطانهای مختلف پرداخته است و تعداد زیادی داده خام با اطلاعات کامل پزشکی و اطلاعات آزمایشگاهی آنها در این بانک موجود میباشد، آدرس دسترسی به این پایگاه tcga-data.nci.nih.gov/tcga میباشد.
در تمام پایگاههای ذکر شده دادههای NGS همراه با آدرس دسترسی به مقاله مرتبط به آن پژوهش وجود دارد، اکثر دادها با فرمت FASTQ موجود میباشند و کاربر میتواند با دانلود این دادها توسط ابزارهای مختلف به آنالیز این دادها بپردازد یکی از نرم افزارهایی که کار آنالیز دادهای NGS را راحت کرده است نرم افزار CLC میباشد. در مرحله اول آنالیز این دادها باید کیفیت دیتا چک شود و معمولا به ازای کل خوانش ها و به ازای هر پوزیشن کنترل کیفیت انجام میشود. در مرحله بعد باید توالی آداپتورها که هنگام توالییابی به توالیها اضافه شدهاند حذف شوند. بعد از نرمال کردن نمونهها مپینگ با رفرنس را انجام میدهیم و بعد از انجام این مراحل میتوانیم توسط الگوریتمهای مختلف واریانتها را چک کنیم و کارهای متنوع دیگری را که میخواهیم انجام دهیم را با الگوریتمها و ابزارهای مختلف انجام دهیم.
هم اکنون پروژه ژنوم اغلب موجودات مدل و انسان به اتمام رسیده است و هر ساله چند پروژه ژنوم دیگر به اتمام میرسد. همگام با انجام پروژههای ژنوم، پایگاههای اطلاعاتی و نرم افزارهای متعددی جهت در دسترسی قرار دادن این اطلاعات به شیوهای مناسب و سریع و بهطور رایگان طراحی و راهاندازی شدهاند. به این منظور مراکزی تحت عنوان
Genome Warehouse راهاندازی شدند تا کلیه اطلاعات موجود اعم از نقشه، توالی، فنوتیپ، علائم بیماری، متون (مقالات، گزارشها و…) و هر گونه اطلاعات ژنوتیپی و فنوتیپی را جمعآوری نموده و با هم مرتبط سازند که در فصلهای آینده با Genome Warehouseها آشنا خواهید شد. همچنین این اطلاعات بایستی قابل بازیابی و قابل درک باشند. به کلیه این مراحل قابل نمایش یا Visualization اطلاعات میگویند.
۳-۱-۲ تفسیر ژنوم (مستندسازی) Genome Annotation
۴۱-ژنومیکس
قبل از ارائه توالیها به بانکهای اطلاعاتی پژوهشگران توالیها را مورد تجزیه و تحلیل قرار میدهند. فرایند مستندسازی شامل دو مرحله پیشبینی ژن و بررسی عملکرد میباشد.
بررسی عملکردی:
ابتدا ساختار ژنی توسط برنامههای پیشبینی کننده اگزونی ab initio مانند GeneScanو Fgenes H پیشبینی میشود (در بیوانفورماتیک ab initio یک روش برای پیشبینی در مورد ویژگیهای بیولوژیکی با استفاده از تنها یک مدل محاسباتی میباشد)¬ سپس توسط BLAST تایید میشوند ¬ ژنهای پیشبینی شده با توالیهای EST و cDNA مقایسه میشوند. (توسط برنامههایی مثل Spidey SIM4 و EST2 Genome و (Gene Wise ¬ پیشبینی توسط یک فرد ماهر بررسی میشوند ¬
بعد از تعیین ORFها بررسیهای عملکردی توسط BLAST و یا جستوجوی موتیفهای اختصاصی توسط PFam و InterPro انجام میشود.
همانطور که شرح داده شد دو راه اساسی برای بررسی عملکرد در دسترس است، راه حل اول استفاده از جستوجوی BLAST میباشد و راه حل دوم استفاده از جستوجوی موتیفهای اختصاصی خانوادههای پروتئینی میباشد. راه اول متکی بر دادههای موجود در پایگاه توالیهای اولیه مانند GenBank است که با دو مشکل روبهرو است: مشکل اول ناکافی بودن اطلاعات در این پایگاهها. بهطور مثال، هنگامی که پروژه ژنوم گیاه مدل Arabidopsis به انتها رسید، تنها ۹ درصد اطلاعات آن با توالیهای موجود در GenBank مشابهت داشت. برای حل این مشکل از روشهای آزمایشگاهی و نرمافزاری متعددی استفاده شده است که در فصل ترانسکریپتومیکس تشریح خواهند شد. مشکل دوم زائد و تکراری بودن رکوردهای GenBank، میباشد که برای حل این مشکل، پایگاه NCBI اقدام به ساخت سه پایگاه UniGene، RefSeq و
Entrez Gene نمود. در این پایگاهها، هم دادهها غیر تکراری هستند و هم تجمعی از دادههای مختلف مربوط به یک ژن یا لوکوس آورده میشود. جرئیات بیشتری در مورد این پایگاهها در فصل بعد آورده شده است برای مثال Uni Gene براساس ترنسکریپتهایی برای یک لکوس یکسان حاصل از دادههای مختلف مثل ESTها تشکیل شده است. راه دوم متکی بر دادههای موجود در پایگاه توالیهای ثانویه است. در واقع، وقتی جستوجو بر مبنای توالی همولوگ امکانپذیر نیست، جستوجوی موتیفها میتواند ما را به این که توالی مجهول مربوط به کدام خانواده پروتئینی است رهنمون باشد.
پیشبینی ژن:
روشها و راهبردهای مختلفی در پیشبینی ژنها وجود دارد که عمده ترین آنها به شرح زیر میباشند.
۱: روشهای آزمایشگاهی.
از جمله روشهای آزمایشگاهی پیشبینی ژن دورگهگیری mRNA و cDNA میباشد که، بیشتر توسط روش نودرن بلات انجام میشده است و هماکنون نیز رواج دارد اما در دهههای اخیر بررسی RNAها تحت عنوان پروژههای EST انجام میشود.
۲: روشهای غیر آزمایشگاهی.
۴۲-فصل دوم
از جمله این روشها میتوان به شناسایی ORFها توسط نرم افزارهای مختلف اشاره کرد. از روشهای دیگر میتوان به شناسایی نواحی رمز کننده با استفاده از ابزارهای بررسی کننده تشابه مثل BLASTX نام برد. روشهای آماری مختلفی هم برای شناسایی ORFها وجود دارند که مهمترین آنها شبکههای عصبی و مدل مخفی مارکوف است. همچنین در رابطه با پیدا کردن ساختار اینترون و اگزون سه راهبرد وجود دارد که به نامهای “روشهای مبتنی بر محتوا”، “روشهای مبتنی بر جایگاه” و “روشهای مقایسهای” شناخته میشوند.
برنامههای پیشبینی ژن:
بهطور کلی برنامههای کنونی پیشبینی ژن در دو دسته اصلی با رویکردهای مبتنی بر ab initio و مبتنی بر همولوژی تقسیمبندی شدهاند. در رویکرد ab initio ژنها را تنها بر اساس توالیهای خاص پیشبینی میکند. اساسیترین موارد، توجه به سیگنالهای ژنی است که شامل کدون های شروع و خاتمه، سیگنالهای قطع اینترون، مکانهای اتصال فاکتورهای رونویسی، مکانهای اتصال ریبوزومی و مکانهای پلی آدنیلاسیون میباشند. دومین خصوصیت که توسط رویکرد ab initio استفاده میگردد شاخصههای آماری میباشد بهطوری که میتوان گفت ترکیب نوکلئوتیدی و الگوهای آماری مناطق کد کننده تمایل دارند که نسبت به مناطق غیرکدکننده به مقدار قابل توجهی متغیر باشند. این شاخصه منحصر به فرد را میتوان با به کار بردن مدلهای احتمالی چون مدل مارکوف (HMM) که به تشخیص مناطق کد کننده از مناطق غیر کد کننده کمک میکنند شناسایی نمود. مدل مبتنی بر هومولوژی پیشبینیها را بر اساس جورشدگی معنا دار با توالی مورد جستوجو انجام میدهد. به عنوان مثال اگر یک توالی ترجمه شده DNA شبیه به یک پروتئین شناخته شده باشد این میتواند یک شاهد قوی بر این مدعا باشد که این قسمت ناحیه کد کننده یک پروتئین است. از ابزارهای پیشبینی ژن میتوان به GENSCAN GENEWISE، GeneID، FGENEH، GRAIL و GeneMar اشاره کرد.
برنامههای مبتنی بر ab initio:
هدف برنامههای پیشبینی ژن ab initio این است که اگزونها را از توالیهای غیرکدکننده تمایز دهند و سپس اگزونها را با ترتیب صحیح به یکدیگر متصل کنند. پیشبینی اگزونها متکی بر سیگنالهای ژن و محتوی ژن است. علاوه بر HMM که قبلا اشاره شد الگوریتمهای مبتنی بر شبکههای عصبی نیز در زمینه پیشبینی ژن رایج هستند. شبکههای عصبی شبیه به سیستم عصبی بیولوژیک است و حاوی متغییرها و گرههایی است که به وسیله توابع وزندهی که آنالوگ سیناپسها هستند به هم متصل میگردند. ویژگی این مدل توانایی آن در یادگیری است. شبکه قادر است که اطلاعات را پردازش نماید و پارامترهای توابع وزندهی بین متغییرها را در طول مرحله ارتقا تغییر داده و پیشبینیهایی را انجام دهد.
الگوریتم شبکههای عصبی در پیشبینی ژن یک شبکه نورال چندین لایه ایجاد میکنند. لایههای ورودی، خروجی و مخفی. ورودی شامل توالی ژن با سیگنالهای اینترون و اگرون است. خروجی احتمال یک ساختار اگزون میباشد. بین ورودی و خروجی learning اتفاق می افتد و ممکن است یک یا چندین لایه مخفی وجود داشته باشد. طی فرایند learning، اطلاعات ساختار ژنی به وسیله شاخصههایی نظیر تکرارهای هگزامری، محلهای برش، محتوی GC ارزیابی و انتقال مییابد. برنامههای ab initio از شبکههای نورال، HMM، و سایر برنامههای مترقی مثل GDA، LDA استفاده میکنند.
۴۳-ژنومیکس
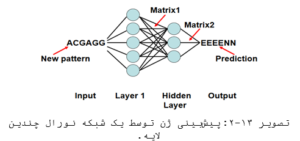
GRAIL یک برنامه تحت وب میباشد که بر اساس شبکههای نورال میباشد. GENSCAN یک برنامه تحت وب میباشد که پیشبینیهایی براساس HMM درجه پنجم انجام میدهد. برنامه HMMgene یک برنامه تحت وب است که از شاخصهای conditional maximum likelihood برای تشخیص شاخصههای کد کننده از غیرکدکننده استفاده مینماید و سپس یک پیشبینی HMM در مناطق دستهبندی شده اجرا میشود و امتداد آن تا بقیه مناطق کد کننده ژن ادامه مییابد. این برنامه بر اساس الگوریتم هیبرید میباشد که هم از شاخصهای مبتنی بر ab initio و هم از شاخص مبتنی بر هومولوژی استفاده مینماید.
برنامههای مبتنی بر همولوژی:
این برنامهها مقایسه توالی مورد مطالعه با نزدیکترین هومولوگ پروتئینی آن در بانک اطلاعاتی را انجام میدهند. نقص این روش اتکای آن بر حضور همولوگها در بانک اطلاعاتی است که اگر هومولوگی موجود نباشد این روش ناکارآمد خواهد بود. GenomeScan یک ابزار تحت وب است که نتایج پیشبینی GENSCAN را با ابزارBLASTX تلفیق مینماید. برنامه EST2genome یک برنامه تحت وب میباشد که یک توالی EST را با یک توالی DNA ژنومی که حاوی ژن مربوطه است مقایسه مینماید. توانایی این ابزار در شناسایی اگزونهای بسیار کوچک و اگزونهای بریده شده است.
برنامههای مبتنی بر Consensus:
چون برنامههای مختلف پیشبینی ژن سطوح متفاوتی از حساسیت و اختصاصیت را دارند پس مطلوب است نتایج حاصل از چندین برنامه را با هم تلفیق کنیم که باعث ایجاد الگوریتمهایی مبتنی بر consensus شده است.
GeneComber یک برنامه تحت وب است که نتایج پیشبینی حاصل از Genescan و HMMgene را با هم تلفیق مینمایند. DIGIT از سه برنامه ab initio یعنی FGENESH، GENESCAN و HMMgene استفاده میکند. این برنامه در ابتدا تمام اگزونهای حاصل از این سه برنامه را گردآوری نموده و ORFها را براساس نمرات مرتب تعیین مینماید.
۴۴-فصل دوم
پیشبینی ژن در پروکاریوتها:
پروکاریوتها ژنوم کوچکی دارند و تراکم ژنی در ژنوم آنها بالاست و توالیهای تکراری بسیار کمی دارند. وجود کدونهای شروع روی DNA شاخص شفافی از محل شروع نسخه برداری نخواهد بود. برای کمک به شناسایی کدون شروع شاخصهای دیگری که مرتبط با ترجمه هستند نیز مورد استفاده قرار میگیرند که یک مورد از آنها توالی شاین دلگارنو است که یک توالی غنی از پورین و مکمل ۱۶Sr-RNA در ریبوزوم میباشد. در انتهای منطقه کدکننده پروتئین و در انتهای هر اپرون یک منطقه خاتمه وجود دارد که میتوانند در پیشبینی ژن کمک نمایند. همچنین برای تشخیص ژنهای پروکاریوتی باید ORFها را شناسایی کرد. همچنین با توجه به این که به ازای هر ۲۰ کدون یک کدون خاتمه به صورت رندوم در یک منطقه غیر کد کننده ژن واقع شده است، لذا یک قالب بلندتر از ۲۰ کدون (بدون یک کدون توقف) میتواند به عنوان یک منطقه کد کننده ژن پیشنهاد گردد، اگر چه حد آستانه برای ORF بهطور معمول یک مجموعه کدونی بزرگتر از ۵۰ یا ۶۰ میباشد. قالب مورد توافق سپس به وسیله حضور سیگنالهای دیگری چون کدون شروع یا توالی شاین- دلگارنو تایید میگردد. یک ORF را میتوانیم در بانکهای پروتئینی جستوجو کنیم و اگر پروتئین همولوگ پیدا کردیم با اطمینان بیشتر میتوانیم بگوییم ORF مورد نظر یک قالب کد کننده پروتئین است.
مدلهای مارکوف میتوانند در پیشبینی آماری یک ژن بسیار مناسب باشند. در مدل مارکوف احتمال یک موقعیت خاص توالی به موقعیت قبلی k بستگی دارد. در اینجا، k درجه مدل مارکوف است. در مدل مارکوف درجه صفر نشان دهنده وقوع هر باز بهطور مستقل از بقیه میباشد. در مدل مارکوف درجه اول وقوع هر باز به باز قبلی آن بستگی دارد و در مدل مارکوف درجه دوم به دو باز قبلی نگاه میکنند که این بیشتر شاخصههای کدونها در یک توالی کد کننده میباشد. هرچه واحد اولیگومر بلندتر باشد به احتمال بیشتری توالی مربوط به یک ناحیه کدکننده میباشد. هرچه درجه مدل مارکوف بزرگتر باشد با صحت بیشتری میتواند ژن را پیشبینی نماید. در توالیهای ژنی کوتاه از یک مدل مارکوف با طول متغییر به نام IMM استفاده میشود و انعطافپذیری بیشتری نسبت به سایر مدلهای مارکوف دارد. از مدلهای با درجات بالاتر معمولاً زمانی استفاده میکنیم که توالی بلند باشد و از مدلهای با درجات پایین زمانی استفاده میکنیم که توالی کوتاه باشد.
نرم افزار GeneMark از برنامههای پیشبینی ژن بر اساس HMMدرجه پنج است. برای توالی مربوط به ارگانیسمی که هنوز مشخص نشده است نزدیکترین ارگانیسم به آن میتواند به عنوان پایهای برای محاسبه مورد استفاده قرار بگیرد. نوعی از GeneMark برای توالیهای یوکاریوتی هم وجود دارد. Glimmer یک برنامه UNIX از TIGER است که از الگوریتم IMM برای پیشبینی مناطق بالقوه کدکننده استفاده مینماید. FGENESB یه برنامه تحت وب است که براساس HMM درجه پنج کار میکند.
یک روش دیگر به نام TESTCODE این حقیقت را استنتاج میکند که بازهای Wobble در منطقه کد کننده تمایل به تکرار دارند، لذا با الگوهای تکراری نوکلئوتیدها میتوان مناطق کدکننده و غیرکدکننده را تمایز داد.
۴۵-ژنومیکس
پیشبینی ژن در یوکاریوتها:
ژنوم هسته یوکاریوتها بسیار بزرگتر از پروکاریوتها و با تراکم ژنی کم میباشد. حد فاصل ژنها بسیار بزرگ بوده و غنی از توالیهای تکراری عناصر قابل جابهجایی (ترنسپوزونها) است. بحث اصلی در تشخیص ژنهای یوکاریوتی تشخیص اگزونها، اینترونها، و محلهای برش است. چندین شاخصه حفاظت شده در ژنهای یوکاریوتی وجود دارند که میتوان با استفاده از آنها پیشبینی محاسباتی کرد، به عنوان مثال برش و اتصال اینترونها و اگزونها به وسیله قانون GT-AG انجام میگیرد که در انتهای اینترون توالی GTAAGTو در انتهای توالی (py)12NCAG وجود دارد. بیشتر ژنهای مهره داران یک توالی منحصر به فرد به نام کزاک به شرح CCGCCATGG دارند. علاوه بر این بیشتر این ژنها تراکمی از توالی CpG دارند که به شناسایی محل شروع نسخهبرداری ژن یوکاریوتی کمک میکنند. سیگنال poly-A نیز به تعیین محل توالی کدکننده انتهایی کمک میکند.
پیشبینی و تجزیه و تحلیل راهاندازها و عناصر تنظیمی:
ارتباط معنیداری بین شناسایی ژن با پیشبینی راهانداز وجود دارد، چنان که اگر یک راهانداز به درستی پیشبینی شود مرزهای ژن به خوبی تعیین خواهد شد و کمک زیادی به پیشبینی ژن خواهد کرد و همچنین اگر یک ژن به خوبی پیشبینی شود کمک زیادی برای پیشگویی راهاندازها خواهد کرد. با توجه به این که تجزیه و تحلیل راهاندازها و عناصر تنظیمی از دو جنبه پیشبینی نواحی راهانداز در توالی ژنومی و تعیین خصوصیات نواحی راهانداز از راه شناسایی نگارههای متصل به عوامل رونویسی باید مد نظر قرار بگیرد دو نوع الگوریتم برای این موضوع وجود دارد:
۱: الگوریتمهای وابسته به الگو.
شامل روشهایی است که مبتنی بر ab initio بوده و پیشبینی de novo را با اسکن کردن یک توالی انجام میدهد. از این نوع الگوریتمها برای جستوجوی توالیهای ژنومی برای شناسایی الگوهای تنظیم کننده شناخته شده، استفاده میشود.
۲:الگوریتمهای وابسته به توالی.
شامل روشهای مبتنی بر تشابه(هومولوژی) میباشد که پیشبینی را بر اساس همردیفی توالیهای هومولوگ انجام میدهند و همچنین روشهای مبتنی بر پروفایل بیان ژن میباشند که از پروفایل بیان ژنهایی که با یکدیگر در همان موجود بیان میشوند استفاده میکنند. پیشبینی مبتنی بر تشابه را انگشت نگاری فیلوژنتیکی نیز مینامند. از الگوریتمهای وابسته به توالی به منظور کشف الگوهای ناشناخته در گروهی از توالیهایی که از نظر عملکردی به یکدیگر وابسته هستند استفاده میشود.
پیشبینی پروموتر در پروکاریوتها:
یکی از جنبههای ویژه در پیشبینی پروموتر پروکاریوتها تعیین ساختار اپرونها است چون اپرونها دارای یک پروموتر مشترک میباشند. روشهایی برای پیشبینی اپرونهای پروکاریوتی وجود دارند که دقیقترین آنها به وسیله wang و همکارانش ارائه گردید. این روشها بر دو نوع اطلاعات اتکا دارند. جهت ژن و فاصله بین ژنی یک جفت از ژنهای مورد نظر و linkage بین آنها.
۴۶-فصل دوم
BPROMیک برنامه تحت وب رایج در رابطه با پیشگویی پروموترهای یوکاریوتی است و نرم افزار FindTermیک برنامه برای جستوجوی سیگنالهای خاتمه دهنده باکتریایی مستقل از “رو” که در انتهای اپرون قرار گرفتهاند میباشند.
پیشبینی پروموتر در یوکاریوتها:
روش ab initio برای پیشبینی پروموترها و عناصر تنظیمی در یوکاریوتها بر جور شدن الگوهای مشترک پروموترها و عناصر تنظیمی شناخته شده اتکا دارد. الگوهای consensus از محلهای اتصال به DNA که بهطور تجربی تعیین شدهاند به دست میآیند. این مکانهای اتصال در پروفایلها جمعآوری شده و در یک بانک اطلاعاتی ذخیره شدهاند. برای افزایش اختصاصیت پیشبینی، از شاخصه منحصر به فرد CpG یوکاریوتی استفاده شده است. با تشخیص جزایر CpG میتوانیم بلافاصله در جزایر بالادست این مناطق به دنبال پروموتر باشیم. CpGProD یک برنامه تحت وب میباشد که پروموترهای حاوی تراکم بالایی از جزایر CpG در توالیهای ژنوم پستانداران هستند را شناسایی میکنند. Eponine یک برنامه تحت وب است که محلهای شروع نسخهبرداری را براساس یک سری از PSSMهایی که از قبل به وسیله چندین محل تنظیمی نظیر جعبه TATA، جعبه CCAAT و جزایر CpG ایجاد شده است پیشبینی مینماید. Cluster-Buster یک برنامه تحت وب مبتنی بر HMM است که جهت یافتن دستههایی از محلهای اتصال تنظیمی طراحی شده است. McPromoter یک برنامه تحت وب است که از یک شبکه نورال جهت پیشبینی پروموتر استفاده مینماید.
روشهای فیلوژنتیک بر اساس Footprinting:
تشخیص عناصر محافظت شده غیرکدکننده DNA که نقشهای حیاتیای را بر عهده دارند Phylogenetic footprinting میگویند و این عناصر را phylogenetic footprints مینامند. این روش میتواند هم در پروکاریوتها و هم یوکاریوتها استفاده گردد. در این روش به فاصله تکاملی در مقایسه توالی منطقه غیرکدکننده در فرادست ژنها توجه میشود که باعث میگردد از بروز مثبتهای کاذب پرهیز شود.
ConSite یک سرور تحت وب است که عناصر عمومی پروموتر را از طریق مقایسه دو توالی اورتولوگ شناسایی مینماید. rVISTA یک ابزار مشابه مقایسه بین گونهای برای شناسایی پروموتر است. PromH یک برنامه تحت وب که محلهای تنظیمی را با استفاده از مقایسه توالی دو به دو شناسایی مینماید. Bayesaligner یک برنامه footprinting و تحت وب است. این برنامه دو توالی را با استفاده از الگوریتم Bayesian که یک روش مقایسه توالی منحصر به فرد است مقایسه مینماید.
روشهای مبتنی بر پروفایل بیان:
۴۷-ژنومیکس
ژنهایی که در بررسی میکرواری پروفایل بیانی مشابهای داشته باشند بهطور هم زمان بیان میگردند. به نظر میرسد علت بیان هم زمان این ژنها اشتراک پروموتر و یا عناصر تنظیمی میباشد. توالی بالادست ژنهایی که بیان هم زمان دارند را میتوانیم با یکدیگر مقایسه کنیم تا یک عنصر تنظیمی مشترک را که به وسیله فاکتورهای نسخه برداری خاص قابل تشخیص هستند را معین کنیم.
MEME یک برنامه مبتنی بر EM است. همانطور که در فصلهای بعد خواهید خواند EM یک روش برای یافتن موتیفهای پروتئینی است اما این روش میتواند برای یافتن موتیف DNA نیز استفاده شود. AlignACE یک برنامه تحت وب است که از الگوریتم نمونهگیری Gibbs جهت یافتن موتیفهای مشترک استفاده مینماید. Melina یک برنامه تحت وب است که بهطور هم زمان از چهار الگوریتم منفرد MEME، Gibbs، Consensus و Coresearch استفاده مینماید. INCLUSive یک ابزار مناسب شبکه است که برای تسهیل نمودن فرایند تجمیع دادههای microarray و یافتن موتیف توالی طراحی شده است.
پایگاه RefSeq
این پایگاه حاوی اطلاعات مرجع غیرتکراری ژنومی، ژنی و پروتئین حاصل مربوط به برخی از موجودات مهم است. حتی در برخی موارد فهرستی از موتیفهای موجود در پروتئین نیز آورده میشود. هم اکنون، دادههای این پایگاه به عنوان قسمتی از یک رکورد Entrez Gene ارائه میشود.
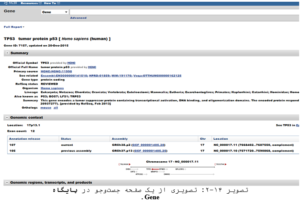
پایگاه Gene
هر رکورد این پایگاه مخزنی از کلیه اطلاعات مربوط به یک لوکوس است. نگاهی به فهرست مطالب (Table of Contebnts) نشان میدهد این اطلاعات شامل توالیهای ژنومی، ژنی و پروتئین، مقالات، نشانگرها، توالی مرجع، بیان ژن، موقعیت نقشهای یا لوکوس، موتیفها و اتصال به پایگاههای دیگر حاوی اطلاعات مربوط است (تصویر ۱۴-۲). پیشتر این پایگاه با نماد
Entrez Gene نمایش داده میشد اما حالا با نماد Gene نمایش داده میشود.
۴۸-فصل دوم
طرح ENCODE
تعیین هویت تمامی عناصر وظیفهدار ژنوم به میزان زیادی شناخت ما از حوادث مولکولی موجود در زمینه نمو، سلامتی و بیماری انسانی را توسعه خواهد داد. برای این منظور، در اواخر سال ۲۰۰۳، مؤسسه ملی تحقیق ژنوم انسانی (NHGRI) طرح دایرهالمعارف عناصر DNA (NECODE) را آغاز نمود. ENCODE که پایه آن در دانشگاه کالیفرنیا و سانتاکروز میباشد، یک تلاش مشارکتی است که رهیافتهای آزمایشگاهی و کامپیوتری را با یکدیگر ترکیب میکند تا هر عنصر وظیفهدار موجود در ژنوم انسانی را شناسایی نمایند. محققین اتحادیه، با زمینهها و تجارب متنوع، برای ایجاد و ارزیابی تکنیکها، فناوریها و راهکارهای جدید با عملکرد بالا تشریک مساعی خواهند نمود تا نقصهای موجود در توانایی خود برای شناسایی عناصر وظیفهدار را برطرف کنند. در طی فاز پایلوت، ENCODE حدود ۱% (Mb30) ژنوم انسانی را برای آنالیز دقیق کامپیوتری و تجربی مورد بررسی قرار خواهد داد. این اتحادیه با چالشهای زیادی روبرو است. علاوه بر اندازه بزرگ ژنوم انسانی و ماهیت مرموز بیشتر توالی آن، دانشمندان میبایست از عهده تنوع عملکرد ژنوم که انواع مختلف سلولها و مراحل متفاوت نمو را مشخص میکنند، برآیند. با توجه به پیچیدگی موضوع، واضح است که هیچ رهیافت تجربی واحدی یا یک نوع سلول برای مرور کامل ارتباطات بین توالی، معیاری و فعالیت ژنومی کافی نخواهد بود.
۴-۱-۲ انتولوژی ژن
با جمعآوری اطلاعات توالی و نامگذاری میلیونها ژن مشکل دیگری بروز کرد و آن ناهمگونی واژههای بکار برده شده توسط زیستشناسان مختلف بود. علاوه بر آن، عملکردهای متفاوت برای یک ژن باعث شده بود یک توالی با نامهای متفاوتی ذخیره شود. بنابراین وقت زیادی توسط پژوهشگران برای تطبیق اطلاعات و واژهها صرف میشود. آز آن گذشته، خودکار نمودن و
۴۹-ژنومیکس
رایانهای نمودن بسیاری از دادهپردازیها دچار مشکل میشود. با توجه به توضیحات ذکر شده باید توصیفهای عملکردی پروتئینها استاندارد باشد. این موضوع باعث ایجاد پروژه انتولوژن ژن(GO) شد. علت سردرگمی این میباشد که پژوهشگرانی که بر روی موجودات مختلف کار میکنند تمایل دارند تا از اصطلاحات متفاوت برای یک ژن یا پروتئین استفاده کنند. پایگاه Gene ontology یا AmiGO دائره المعارفی از اطلاعات برای توضیح ژن و محصولات آن است که سعی بر یکنواخت سازی نامگذاریها و واژهشناسیها دارد. توصیف GO سه دسته اطلاعات به ما میدهد که شامل فرآیندهای زیستی، اجزای سلول و عملکرد مولکول میباشد.
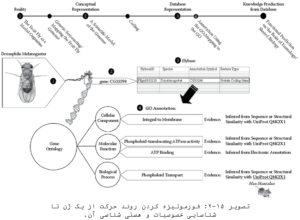
در واقع هر محصول ژنی با توجه به موقعیت سلولی (Cellular component) در یک فرایند زیستی خاص
(Biological process) درگیر بوده و در نتیجه عملکرد خاصی (Molecular function) را در سلول انجام میدهد. برای مثال، سیتوکروم C با توجه به موقعیتهای ماتریکس میتوکندریایی و غشای درونی میتوکندری با عملکرد اکسیدوردوکتازی در فرایندهای زیستی فسفوریلاسیون اکسیداتیو و مرگ سلولی درگیر است. با توجه به مفاهیم ذکر شده پروژه Gene Ontology در سال ۱۹۹۸ توسط پژوهشگران مطالعه ژنوم پایهگذاری شد و به عبارتی کنسرسیوم Gene Ontology شکل گرفت. AmiGO در واقع ابزار قدرتمندی برای جستوجو و بازیابی اطلاعات از پایگاه دادهای Gene ontology است. برای ورود به این پایگاه باید از آدرس http://www.geneontology.org/amigo استفاده کرد. برای جستوجو میتوان از نام ژن، محصول ژن، توالی ژن مورد جستوجو و نام GO ژن استفاده کرد.
۵۰-فصل دوم
۵-۱-۲ مستندسازی خودکار
با توجه به میزان زیاد دادههای ژنومی برای مستندسازی روشهای خودکار نیاز است. روش کار براساس شناسایی نواحی دارای همولوژی میباشد. اگر توالی یک ژن یا محصول آن ژن شباهت معنیداری با توالی موجود در پایگاه اطلاعاتی داشته باشد انتقال بررسیهای عملکردی رخ میدهد.
Gene Quiz یک سرور اینترنتی برای مستندسازی یک توالی پروتئین است. قادر به بررسی شباهت توالی مورد تقاضا و مستندهای عملکردی پروتئین بر اساس چندین خصوصیت است. این نرم افزار توالی را با توالیهای درون بانک ها با الگوریتمهای BLAST و FASTA مقایسه میکند. همچنین تجزیه و تحلیلهای دمینها با کمک Blocks و PROSITE انجام میشود.
۶-۱-۲ مستندسازی پروتئینهای فرضی
عملکرد حدود ۴۰% از ژنهایی که در ژنومهای تازه توالییابی شده پیدا شدهاند معلوم نیست و از آنها تنها میتوان به عنوان ژنهایی که رمز کننده پروتئینهای فرضی هستند یاد کرد. شناسایی آزمایشگاهی این پروتئینهای فرضی کاری وقتگیر و پر هزینه است. به این دلیل بررسی را توسط ابزارهایی که همولوژی جزیی را بررسی میکنند انجام میدهند. شناسایی همولوژی جزئی شامل کلاسیفیکیشن دمینهای پروتئین، پیشبینی ساختار دوم و سوم، مکان سلولی پروتئین و اینترکشن پروتئین- پروتئین میباشد.
۷-۱-۲ ساختار ژنوم
تجزیه و تحلیل ساختار ژنوم در موجودات مختلف توسط اندازهگیریهای آماری انجام میشود. تجزیه و تحلیل ساختار ژنوم شامل اندازه، ترکیب نوکلئوتیدها، فراوانی رمزهای ژنتیک، تعیین نواحی حفاظت شده میباشد. فراوانی GC و AT بین موجودات مختلف متفاوت است. محتوی GC و AT در جریان تکامل دچار تغییرات اساسی شده است. همچنین رمزهای اسید آمینههای مشابه در موجودات مختلف یکسان نیست. تحقیقات نشان داده است که سازماندهی ژنوم انسان و موش مشابه است. براس تشریح تشابه بین قطعات کروموزومی اصطلاحات متعددی تعریف شده است.
ژنهای سینتنیک: چنانچه دو یا چند ژن بر روی یک کروموزوم قرار داشته باشند احتمالاً این ژنها به هم پیوسته هستند و سینتنیک میباشند.
سینتنی: اگر ژنهای سینتنیک پروتئینهای ارتولوگ در یک کروموزوم منفرد در گونههای مخلتف حفظ شده باشد از اصطلاح سینتینی استفاده میشود. ترتیب ژنها در کروموزومها مورد توجه نیست.
لینکاژ: اگر ترتیب ژنها در کروموزومها حفظ شده باشد به آن نواحی قطعات حفظ شده یا لینکاژ گویند.
۸-۱-۲ گروههای پروتئینی ارتولوگ
با کامل شدن پروژههای توالییابی ژنومهای مختلف توجه به تجزیه و تحلیل و گروهبندی ژنهای پیشبینی شده و توجه به عملکرد آنها بیشتر شده. با توجه به این که مقایسه کل ژنوم یا پروتئوم بسیار سخت است بستههای تجاری ۵۱(مثل برنامه Gen Light) ایجاد شده است که امکان مقایسه مجموعه دادههای توالیهای بزرگ را میدهد.
۵۱-ژنومیکس
روش خوشهبندی دیگر برای گروهبندی پروتئینها، تولید خوشههای پروتئینی ارتولوگ (COG) است. از نظر تکامل همه پروتئینهای داخل یک COG از یک جد مشترک اولیه یا فرایند گونهزایی یا مضاعفشدگی به وجود آمدهاند. تولید COG ها به وسیله مقایسه جفتی توالی همه پروتئینهای مورد مطالعه و تجزیه و تحلیلهای بعدی شبکه روابط حاصله صورت میگیرد. تعیین پروتئین های ارتولوگ در یک گروه از گونهها از مهمترین کارهای بررسی تکامل و شناسایی عملکرد پروتئینهای ناشناخته است.
سیستمهای پیچیدهای برای گروهبندی پروتئین های ارتولوگ به وجود آمدهاند که یکی از بهترینها COG در NCBI است. در حال حاضر در این پایگاه اطلاعاتی ۹۷۲۴ خوشه پروتئین ارتولوگ از ۷۳ ژنوم که توالییابی کامل شدهاند وجود دارد. پایگاه MBGD محاسبه پویایی خوشهها را طبق پارامترهای تنظیم شده به وسیله کاربر تسهیل میکند.
COGs یک سیستم طبیعی خانوادههای ژنی از ژنومهای کامل است. بهطوریکه خوشههای گروههای ارتولوگ به وسیلهی مقایسه توالی پروتئینی در ۴۳ ژنوم کامل رسم شده است. از آنجا که هر COG شامل پروتئینهای خاص یا گروهی از پارالوگها مربوط به حداقل ۳ دودمان هستند، بنابراین با دمین حفاظت شده قدیمی تطابق دارند.
۹-۱-۲ نواحی رمز کننده
نواحی غیر رمز کننده در پروکاریوتها کم میباشد و شناسایی و تعیین ژنها در پروکاریوتها به صورت مقایسهای آسان است. بیش از ۸۰ درصد ژنوم پروکاریوتها رمزکننده پروتئین و RNA است. نواحی غیررمز شده در یوکاریوتها زیاد است و شناسایی ژنها دشوار است. همچنین فرایند پردازش و وجود اینترونها که باعث میشوند انواع مختلفی پروتئین ایجاد شود کار را دشوارتر میکند.
۱۰-۱-۲ نواحی غیررمز کننده
نواحی غیر رمز کننده از این لحاظ که میتوانند در تنظیم نواحی ژنومی تاثیرگذار باشند مهم میباشند. زمانی که دو ژنوم خیلی بزرگ نزدیک بههم با همدیگر مقایسه و مشابهتهایی بین نواحی غیررمزکنندیشان پیدا میشود احتمال شناسایی حوزههای تنظیمی بیشتر میشود.
۱۱-۱-۲ تعداد ژنها در ژنوم
تعداد ژنها در انسان در حال بررسی میباشد و کار پیچیدهای است. در ابتدا تصور بر این بود که حدود ۱۲۰ هزار ژن در ژنوم انسان وجود دارد. بعد از توالییابی کامل و با استفاده از ابزارهای مشخص کننده ژنها، تعداد ژنهای انسان بین ۲۵ تا ۳۰ هزار عدد برآورد شده. تا به حال تعداد دقیق ژنهای انسان مشخص نشده است و بعضی از پژوهشها تعداد ژنهای انسانی را ۱۸ هزار عدد اعلام میکنند. تعداد ژنهای ژنوم برنج دو برابر ژنوم انسان است که این دیدگاه را که انسان گونه غالب زمین است را به چالش کشیده است. اما باید توجه کرد پیچیدگی ژنوم را نمیتوان از راه تعداد ژنها توجیه و بررسی کرد.
۵۲-فصل دوم
۱۲-۱-۲ اقتصاد ژنوم
با استفاده از آنالیز ESTها مشخص شده در حدود ۱۰۰ هزار پروتئین در انسان بیان میشود. در حالی که بررسیهای ژنوم حدود ۳۰ هزار ژن را مشخص کرده است. ساز و کار پیشبینی شده بسیار پیچیده میباشد و برای بیوانفورماتیک یک چالش محسوب میشود. ساز و کار اصلی مسئول تنوع پروتئین، پردازش متفاوت است. سنتز پروتئینهای بیشتر با وجود تعداد کمی ژن، یکی از راهبردهای بسیار مهم است که موجودات یوکاریوتی برای رسیدن به حداکثر تنوع فنوتیپی از آن استفاده میکنند. اگزونهای متفاوت به صورت متفاوت به هم متصل میشوند و پروتئینهای مختلف ایجاد میکنند. سازوکار دیگری همچون اتصال اگزونهای متفاوت تحت عنوان Exon Shuffling (برخورد اگزونی)وجود دارد که در این ساز و کار دوم اگزونهای مختلف از ژنهای مختلف به یکدیگر متصل میشوند. از سایر ساز و کارها میتوان پردازش ترانس و پدیده ژن داخل ژن را نام برد. حدود ۶۶% ژنهای انسان در هنگام بیان، پدیدههای پردازش متفاوت و برخورد اگزونی را نشان میدهند و بیش از ۹۰% کل پروتئینها را تولید میکنند. در مگس سرکه ژن DSCAM، ۱۱۵ اگزون دارد و با پردازش متفاوت ۳۸ هزار پروتئین متفاوت تولید میکند. این توانایی بسیار بالا برای تولید پروتئینهای متفاوت، پیچیدگی واقعی یک ژنوم محسوب میشود نه تعداد ژنها. پایگاههای اطلاعاتی Prosplicer پایگاه اطلاعاتی نسخههای حاصل از پردازش متفاوت ژنهای انسان است. پردازشهای متفاوت یک ژن با استفاده از برنامههای SIM4 و TBLASN شناسایی میشوند.
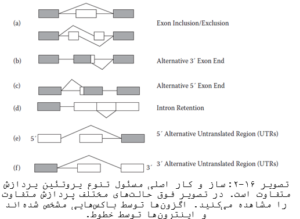
۵۳-ژنومیکس
۲-۲ ژنومیکس مقایسهای
مقایسه کل ژنومهای موجودات مختلف با یکدیگر را ژنومیکس مقایسهای مینامند. ژنومیکس مقایسهای شامل مقایسه تعداد ژن، محل ژن و محتوی ژن میباشد. ژنومیکس مقایسهای به شناسایی مناطق حفظ شده بین ژنومها کمک میکند. میتوان اطلاعاتی درباره ساز و کار تکامل ژنوم و انتقال افقی ژن بین ژنومهای مختلف به دست آورد و همچنین ژنومیکس مقایسهای به ما در مهندسی مسیرهای متابولیسمی کمک میکند.
مباحثی که در ژنومیکس مقایسهای مورد بحث قرار میگیرند به پنج دسته زیر تقسیم میشوند:
۱) همردیفی کل ژنومها
۲) انتقال افقی ژنها
۳) روش درون ژنومی
۴) مقایسه ترتیب ژنها بین دو موجود
۵) ترسیم ژنومهای حداقل
۱-۲-۲ همردیفی کل ژنومها
با افزایش تعداد ژنومهای توالییابی شده میتوان توالیهای حفظ شده بین ژنومها را که به مشخص شدن حضور عناصر عملکردی حفظ شده کمک میکند، به وسیله هم ردیفی کل ژنومها با یکدیگر شناسایی کرد. برنامههای همردیفی معمولی برای مقایسه توالیهای بسیار بزرگ استفاده نمیشود و همچنین و به علت طویل بودن توالی نمایش نتیجه چالش برانگیز است. (بحث همردیفی در فصل ششم به صورت تفصیلی آمده است).
MVMmer یک ابزار در پایگاه TIGR میباشد که برای هم ردیفی دو توالی ژنومی کامل و مقایسه مکان ارتولوگها به کار میرود، همردیفی کل ژنوم به صورت پلات نقطهای که با خطوطی از نقطهها به هم وصل شدهاند نمایش داده میشود. این برنامه شکل تغییر یافته BLAST است.
BLASTZ یک ابزار تغییر شکل یافته BLAST است که بعد از یافتن نواحی همردیفی شده با یک روش وزندهی و همچنین با تغییرات حداقل همردیفی را نمایش میدهد.
LAGAN ابزاری است که ابتدا نواحی کوتاه که کامل جفت میشوند را پیدا میکند در ادامه هم ردیفی با الگوریتم نیدلمن وانچ انجام میشود.
Pip Marker ابزاری است که با استفاده از روش تجربی BLASTZ نواحی مشابه را پیدا میکند.
MAVID ابزاری است که براساس الگوریتم پیشرونده Clustal عمل میکند. لنگرگاههای بین توالی با الگوریتم اسمیت واترمن انجام میشود.
Genom Vista یک برنامه جوستجوگر در ژنوم انسان، موش، موش صحرایی و دروزوفیلا است. از برنامهای به نام BLAT برای پیدا کردن لنگرگاهها استفاده میشود. سپس همردیفی را از نواحی لنگرگاه توسط برنامه AVID ادامه میدهد.
۲-۲-۲ انتقال افقی ژن
بیشتر در ژنومهای پروکاریوتی دیده میشود و توسط روشهایی مثل ترانسفورماسیون، کونجوگیشن و ترنسداکشن صورت میپذیرد گروه بندی غیر طبیعی که در درخت فیلوژنتیک رخ میدهد بیانگر امکان انتقال افقی ژنها، بین گونه های مورد بررسی است.
۵۴-فصل دوم
۳-۲-۲ روش درون ژنومی
این روش نواحی از یک ژنوم غیرمعمول را شناسایی میکند. آمارههای نوکلئوتیدی غیرمعمول در نواحی ژنومی به شناسایی ژنهای خارجی در ژنوم کمک میکند. از پارامتر انحراف، GC به عنوان آماره نشان دهنده وجود عناصر ژنتیکی کسب شده استفاده میشود که از طریق فرمول به دست میآید.
ACT برنامهای است که ژنومها را برای بررسی حذف و اضافهشدگی بررسی میکند.
SWaap برنامهای است که نواحی رمزکننده را از نواحی غیررمز کننده جدا میکند و انحراف GC را نشان میدهد.
۴-۲-۲ مقایسه ترتیب ژنها
هنگامی که ترتیب گروهی از ژنها در بین ژنومهای مختلف حفظ شده باشند به این حالت سینتنی گویند اگر روابط سیتنی برای ژنهای معینی در بین پروکاریوتهای منشعب از هم مشاهده شود آنها کلید مهمی برای نشان دادن روابط عملکردی است (مثل اپرونها).
Genorder برنامهای است که مقایسه توالیهای ژنومی را انجام میدهد.
۵-۲-۲ یافتن ژنوم حداقل
تعریف ژنوم حداقل یعنی تعدادی ژن از کل ژنوم که برای سلول یک زندگی حداقلی را فراهم میآورند.
– پیدا کردن ژنومهای حداقل به فهم ژنهای دخیل در مسیرهای متابولیسمی کلیدی که برای حیات سلول ضروریاند کمک میکند.
– در این تجزیه و تحلیل ژنهای ارتولوگ که بین تعدادی از ژنومهای دور از هم مشترکند، شناسایی میشوند.
Coregenes برنامه ای است که هسته های ژنی را بر اساس مقایسه چهار ژنوم کوتاه شناسایی میکند. این برنامه از ابزار Iterativ BLAST برای پیدا کردن ژنهای ارتولوگ استفاده میکند.
۵۵-ژنومیکس
۳-۲ ژنومیکس عملکردی
با تعیین توالی ژنوم، اطلاعات در مورد ژنها و بیان آنها فراهم میشود اما نمیتوان به وظیفه و عمل ژنها پیبرد. از ژنومیکس عملکردی برای بررسی الگوهای متفاوت بیان ژنها در مراحل تکوین و یا محیطی متفاوت استفاده میشود. به دلیل اینکه مقدار پروتئین اغلب از روی میزان mRNA تولید شده در سلول قابل پیشبینی نیست و همچنین تغییرات پس از ترجمهای که در پروتئین رخ میدهد را نیز نمیتوان از روی mRNAها تشخیص داد نتیجه میگیریم مطالعه پروتئینها ضروری میباشد در این راستا پژوهشگران از طریق مطالعه پروتئین ها به تجزیه و تحلیل عملکرد پروتئینها در بافتها میپردازند. روشهایی که در ژنومیک عملکردی استفاده میشوند اطلاعات تعداد زیادی از بانکهای اطلاعاتی را فراهم کردهاند که در این قسمت با برخی از مهمترین روشها همچون ESTها، SAGE (تجزیه ترتیبی بیان ژن)، ریز آرایه، Realtim PCR، مطالعه پروتئین ها، بررسی تغییرات بعد از ترجمه، دستهبندی پروتئینها و جایابی، متابولومیکس، سیستم بیولوژی، تجزیه و تحلیل مقایسهای متابولیک، پایگاه اطلاعاتی مولکولهای شیمیایی آشنا خواهید شد. در فصلهای آینده هر کدام از این مباحث به تفصیل شرح داده میشوند.
۱-۳-۲ ESTها
یکی از بهترین و قابل اعتمادترین راههای شناسایی واحدهای رونویسی، استفاده از روش مشهور به تعیین شناسه توالیهای رونویسی یا EST میباشد. این توالیها کوتاه بوده و از یک یا هر دو انتهای هر همسانه با یکبار توالییابی مشخص میشوند
(در فصل نهم این روش با تفصیل شرح داده شده است). در عمل، با استفاده از آغازگرهای مبتنی بر توالی ناقل اقدام به تعیین توالی انتهاهای قطعه درون یک همسانه از کتابخانه cDNA میشود که بهطور تصادفی برداشته شده است. در صورتی که توالی دارای حداقل طول ۱۰۰ bp با کمتر از ۳ درصد نوکلئوتید نامشخص (N) باشد، آن توالی در پایگاه GenBAnk یا مشابه آن ذخیره میشود.
۲-۳-۲ تجزیه ترتیبی بیان ژن (SAGE)
در این فناوری، قطعات cDNA با برچسبهایی به هم متصل شده و تعیین توالی میشوند. در صورتی که تعداد کافی از برچسبها تعیین توالی شوند، محقق میتواند از نظر کمی میزان یک mRNA ویژه را در یک سلول اندازهگیری کند.
(در فصل نهم این روش با تفصیل شرح داده شده است.)
۳-۳-۲ میکرواری
ریزآرایهها اسلایدهای میکروسکوپی هستند که دارای سریهای مرتبی میباشند. انواع ریزآرایه داریم به نامهای ریزآرایه DNA، ریزآرایه RNA و ریزآرایه پروتئین که این اسامی بستگی به مادهای دارد که روی اسلاید قرار میگیرد. سطح اسلایدها دارای گروههای شیمیایی فعال میباشند که موجب پایدار کردن و پیوند DNA بر روی اسلاید میشوند. فناوری دیگری به نام فتولیتوگرافیک مولکول DNA را بهطور مستقیم بر روی اسلاید میسازد. میتوان توسط فناوری ریزآرایه SNPها را بررسی کرد اما کاربرد اصلی ریزآرایهها تعیین سطح بیان ژن در نمونه است. دادههای میکرواری در سه پایگاه در دسترس هستند در EBI در بخش Array Express در NCBI در بخش GEO و در DDBJ در بخش CIBEX. (در فصل نهم این روش با تفصیل شرح داده شده است.)
۵۶-فصل دوم
۴-۳-۲ Real Time PCR
توسط این روش با دقت و سرعت بالایی در حین واکنش PCR میتوان مقدار DNA ساخته شده را اندازهگیری کرد. در این روش به راحتی میتوان محاسبات کمی را انجام داد. حساسیت این روش ۱۰۰۰ برابر بیشتر از روش هیبریداسیون دات بلات است. PCR زمان واقعی تجمع محصولات را در فاز تصاعدی تعیین میکند. یکی از روشهای پرکاربرد در Real Time PCR براساس استفاده از رنگهای فلورسنس متصل شونده به DNA دو رشتهای است. سه روش دیگر نیز وجود دارد که استفاده از شناساگرهای مولکولی است که به رنگ فلورسانس متصل هستند.
۵-۳-۲ مطالعه پروتئینها
ژنها قطعاتی از DNA هستند که به خودی خود فاقد عملکرد بیوشیمیایی هستند. در فاصله بین تبدیل اطلاعات نهفته در یک ژن به یک عملکرد بیوشیمیایی خاص وقایع مختلف رخ میدهد که سرانجام فعالیت ژن یا پروتئین حاصل، محل ایفای نقش آن و نوع آن را مشخص میکند. بروز یک فنوتیپ در بیشتر حالات محصول یک ژن نیست به ویژه در مورد صفات کمی، اثرات اپیستازی و پلیوتروپیک بین ژنها و همچنین میانکنشهای فراوانی بین پروتئینهای مختلف در تعامل با شرایط محیطی در نهایت یک فنوتیپ خاص را ایجاد میکند. در الکتروفورز دو بعدی نقاطی از ژل (لکههای رنگآمیزی شده) که با ژل کنترل تفاوت دارند (در اندازه لکه یا شدت رنگ) از ژل جدا میشوند و مورد هضم آنزیمی قرار میگیرد (اکثرا توسط تریپسین) محلول پپتیدی حاصل را توسط طیفسنجی جرمی مورد بررسی قرار میدهند و وزنهای پپتیدها به دست میآید. این جرمها را در بانکهای جرمی جستوجو میکنند تا بفهمند لکه مربوط به چه پروتئینی بوده است. (در فصل هشتم این موضوع به همراه بحث پروتئومیک پرداخته شده است.)
۶-۳-۲ بررسی تغییرات بعد از ترجمه
بسیاری از پروتئینها بعد از ترجمه دچار گلیکوزیلاسیون و یا فرایندهای دیگری میشوند که به این فرایندها تغییرات پس از ترجمه میگویند. در تجزیه و تحلیل پروتئومها بررسی تغییرات بعد از ترجمه بسیار مهم است.
۷-۳-۲ دستهبندی و جابهجایی پروتئینها
مطالعه سازوکار انتقال پروتئینها Protein Sorthing نامیده میشود.
۸-۳-۲ متابولومیکس
امروزه بیش از ۲۰۰۰ متابولیت در انسان شناسایی شده. خزانه متابولیکی کل سلول متابولوم نامیده میشود. همانند پروتئومیکس و یا ترنسکریپتومیکس فناوریای وجود ندارد که بتواند کل متابولیتها را یکجا بررسی کند. اسپکتروسکوپی NMR روشی است که اطلاعاتی در خصوص خصوصیات فیزیکی و شیمیایی متابولیتها در اختیار قرار میدهد (در فصل دهم به تفصیل به موضوع متابولومیکس پرداخته شده است.)
۹-۳-۲ سیستم بیولوژی
در سیستم بیولوژی با کمک تلفیق دادههای ژنومیک، پروتئومیک، ترنسکریپتومیکس، متابولومیکس سعی میشود سیستم سلولی شناخته شود و رفتارهای سلول فرمولیزه شود و مدل سلولی طراحی میشود. در سیستم بیولوژی نه تنها به رفتارها و شبکههای داخل سلول توجه میشود بلکه رفتار سلول و ارتباطات آن با سلولهای مجاور و ارتباط سلول با محیط نیر بررسی میشود. هدف بعدی این است که با استفاده از ابزارهای کامپیوتری و الگوریتمهای ریاضیاتی مراحلی از فرایندهای سلول که شناسایی شده است تکمیل شود (مثلا فرایند فاگوسیتوز). در نهایت با فورمولیزه کردن کل فرایندهای سلول به یک سیستم جامع دست خواهیم یافت که درک ما را نسبت به سلول و فرایندهای آن کاملتر خواهد کرد (در فصل چهاردهم به تفصیل به جنبههای مختلف سیستم بیولوژی پرداخته شده است)
۵۷-ژنومیکس
– SBML یک زبان کامپیوتری مبتنی به XML است که در سیستم بیولوژی کاربرد دارد.
– پایگاه اطلاعات Biomodels در EBI حاوی مدلهای کامپیوتری مربوط به سیستم بیولوژی میباشد.
۱۰-۳-۲ تجزیه و تحلیل مقایسهای متابولیکی
برای پیشبینی ژن، تاکید ویژه بر ژنهایی است که در مسیرهای متابولیسمی نقش دارند. با استفاده از پیشبینی ژن میتوان مشخص کرد که آیا این موجود چرخه متابولیکی مورد نظرمان را دارد یا نه. پایگاههای Reactom , Ecocyc , KEGG برای مقایسه متابولومها و چرخههای متابولیسمی کاربرد فراوانی دارند.
۱۱-۳-۲ پایگاه اطلاعاتی مولکولهای شیمیایی
پایگاه Pabchem در NCBI یک پایگاه اطلاعاتی مولکولهای شیمیایی است. که به سه بخش کلی تقسیم میشود Pubchem Substance، Pubchem Compound و Pubchem BioAssoy.